- +1
中疾控等重磅研究揭示新冠病毒演变:突变如何显著增强传染力
当地时间3月2日,中国医学科学院北京协和医院、中国疾控中心、加州大学洛杉矶分校、匹兹堡大学、湖南大学五家合作单位的研究人员共同在生物科学预印本网站bioRxiv在线发表了一项针对新冠病毒演化过程中的突变、重组和插入的重磅研究(“Mutations, Recombination and Insertion in the Evolution of 2019-nCoV”)。
研究通过分析病毒的突变,解释新冠病毒为何传染性能得到显著增强。
团队还进一步估计,大多数人类新冠病毒(2019-nCoV)和蝙蝠RaTG13病毒蛋白之间的差异发生在2005年至2012年之间,而人类SARS病毒和蝙蝠SARS样冠状病毒之间的差异发生在1990至2002年之间。
而除了点突变外,他们认为还有潜在的证据表明重组也是2019-nCoV进化的机制。他们的结果表明,2019-nCoV的S蛋白可能从穿山甲冠状病毒衍生出来,而不是蝙蝠冠状病毒RaTG13。而在2019- nCoV 的进化过程中,RaTG13样冠状病毒和穿山甲样冠状病毒病毒株之间可能发生了重组。
该论文通讯作者为中国疾控中心病毒病预防控制所党委书记武桂珍,中国疾控中心病毒病预防控制所研究员谭文杰,中国医学科学院生物医学大数据中心主任、苏州系统医学研究所所长助理蒋太交,加州大学洛杉矶分校微生物与免疫遗传学系教授程根宏。
研究团队收集并分析了120个2019-nCoV基因组序列,包括11个来自中国患者的新基因组。他们发现,尽管2019-nCoV、人和蝙蝠SARS-CoV(严重急性呼吸综合征冠状病毒)在整体基因组结构中具有高度同源性,但它们通过受体结合域中(RBD)的潜在重组,演变成具有不同受体进入特性的两组病毒。
研究团队发现,2019-nCoV在刺突蛋白(S蛋白)的S1和S2结构域之间存在独特的四个氨基酸插入(PRRA),它可能是一个弗林(Fruin)或TMPRSS2(跨膜丝氨酸蛋白酶2)酶切位点。而此前的研究表明,冠状病毒可能发生蛋白酶裂解,从而触发病毒-细胞膜之间的融合。这种启动和触发融合机制的灵活性极大地调节了不同冠状病毒的致病性和趋向性。
研究团队提出,RBD的潜在重组,以及独特的弗林蛋白酶切位点的存在,可以解释新冠病毒传染性的显著增加。
当前,2019-nCoV在全球已经导致超过77000人感染、2400人死亡(截至论文提交),其基因组在系统发育上与蝙蝠SARS病毒RaTG13株最相似,该病毒株于2013年在中国云南被首次分离。
研究人员表示,截至目前已经有很多针对新冠病毒的研究,但导致这种病毒感染和分子进化的机制仍不清楚,这项研究揭示了乙类冠状病毒的进化、特异性、以及增强感染力的可能机制,为2019-nCoV的进化和传播提供了全面的见解。
他们认为,利用从不同地点、不同时间点的患者身上分离到的2019-nCoV毒株进一步追踪基因组突变,将为理解这种快速传播病毒的分子进化提供思路。
2019-nCoV传播中发生的突变
研究团队将2019-nCoV的感染率与最近暴发的β冠状病毒,即2002年的SARS病毒和2012年的MERS(中东呼吸综合征)病毒的感染率进行了比较。
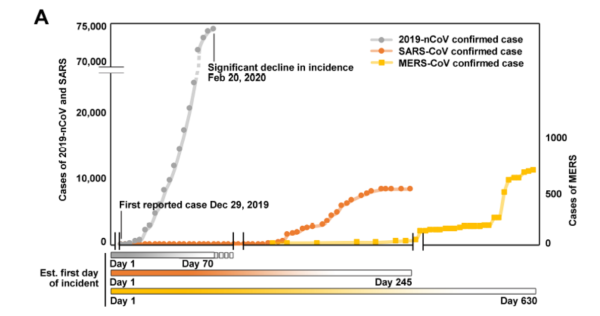
与SARS和MERS相比,2019-nCoV的传播速度要快得多。迄今为止,已确认的2019-nCoV病例比2002年整个SARS暴发期间的病例要多。
在过去的18年中,科学家发表了很多冠状病毒的基因序列,其中包括2002年SARS暴发期间从不同国家分离出的SARS病毒株,从沙特阿拉伯和阿拉伯联合酋长国等中东国家也分离出很多MERS病毒株。
研究团队这次从包括武汉在内的多个中国城市的新患者中收集并测序了11条全长2019-nCoV基因组序列。
系统发育分析表明,与人类SARS、MERS和其他冠状病毒相比,这11个新的2019-nCoV病毒株与其他2019-nCoV病毒株类似,它们与蝙蝠冠状病毒RaTG13病毒株同源性更高。
在氨基酸水平上,它们只在与人类和蝙蝠SARS中相应氨基酸序列相同的一致序列位置上有少量随机突变。
为了鉴定新的遗传突变,研究人员使用新冠病毒株EPI_ISL_402125作为根,为GIAID(全球共享禽流感数据倡议组织)中的所有120个新冠病毒的可用完整基因组构建了系统树(更新至2020年2月18日)。
研究团队发现,根据核苷酸位置8517和27641,2019-nCoV病毒株可分为两个主要类别。
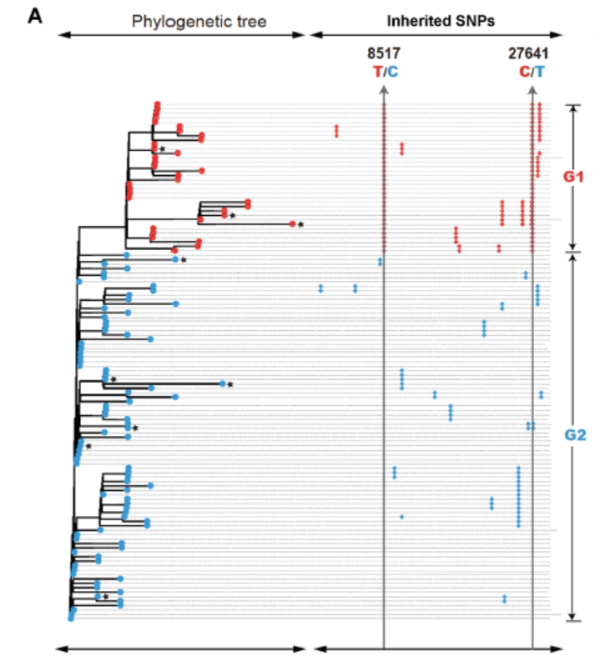
120个2019-nCoV全长基因组的序列比对,包括11个新的基因组(以星号突出显示),G1和G2分别用红色和蓝色标记
第1组(G1)的所有病毒株在8517的位置有胸腺嘧啶,在27641位置有胞嘧啶,这与SARS中相应的核苷酸表现相同;而第2组(G2)则相反,在8517有胞嘧啶,在27641则有胸腺嘧啶。
以上两组病毒的流行病学数据显示,最早的G1病毒株(EPI_ISL_406801)于2020年1月5日在武汉被收集,而最早的G2株则于2019年12月24日在武汉被分离得到。
上述两组基因群在同一城市中的存在表明它们是共循环的,但是在疫情暴发早期它们的进化是趋同的。在每个组中,研究人员还观察到了多个病毒株的其他共有突变。
基于这些潜在的可遗传突变以及识别时间和位置,研究人员生成了一个“突变树”图,以跟踪单个共享突变并显示不同分离株之间的关系。
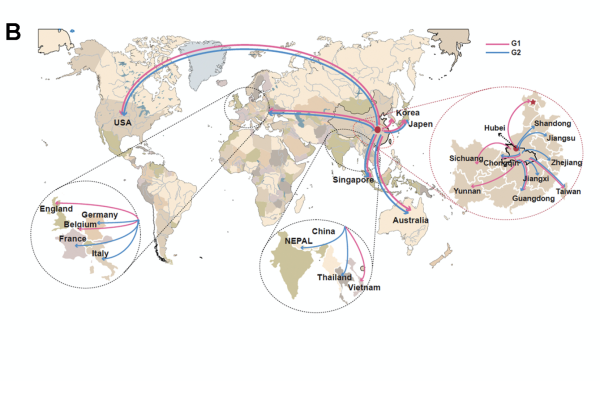
例如,今年1月10日至1月15日在广东省识别出的5个病毒株属于上述第1组病毒,它们在核苷酸位置28578上均有相同的突变,这表明它们可能是由同一人传播的。类似的病毒可能传播给了1月29-1月31日在日本发现的3例病例,它们的病毒在2397核苷酸位置有额外突变,这一类似病毒可能也传播给了1月22日在美国发现的1例病例,这例病例的病毒在10818核苷酸位置有一个额外突变。
研究团队还提到G10818T这个位置非常有趣,因为它在第1组和第2组中均由数个独立的病毒株共享,这导致了orf1ab多蛋白内的L3606F氨基酸突变。
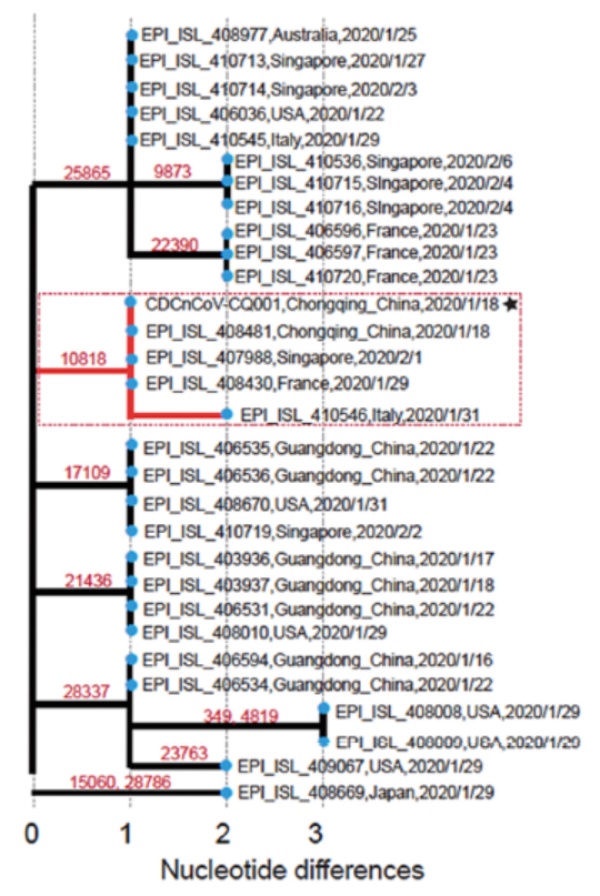
G10818T在第1组和第2组中均由数个独立的病毒株共享
目前尚不清楚第1组和第2组病毒株在10818位点的共同突变是否具有任何生长优势,但穿山甲和蝙蝠冠状病毒在相同位置同样有L3606V突变。
从树状图来看,这两组病毒株都已传播到报告有2019-nCoV病例出现的大多数国家和地区,几乎没有例外,这表明这两组病毒都是可以快速传播的。
研究团队在讨论环节提到,尽管这两个不同的2019-nCoV组是在从动物传播给人类之前或之后进化尚待确定,这两个群体都是首先在武汉被发现,然后传播到中国的不同地区和多个国家。
2019-nCoV与穿山甲冠状病毒的比较
目前与新冠病毒最紧密相关的病毒株为β型冠状病毒RaTG13,RaTG13此前从中华菊头蝠中分离出。研究团队使用核苷酸序列对特定病毒蛋白(例如orf1a,S蛋白,基质和核衣壳)进行了其他系统发育分析后发现,RaTG13病毒株与其他蝙蝠类SARS样冠状病毒株同样有密切关系。
研究人员进一步估计,大多数2019-nCoV和RaTG13病毒蛋白之间的差异发生在2005年至2012年之间,而人类SARS病毒和蝙蝠SARS样冠状病毒之间的差异发生在1990至2002年之间。
研究人员在讨论环节总结道到,“我们的进化时钟分析估计,2019-nCoV分别于大约12年前和30年前从RaTG13和人类SARS-CoV中分化出来。”而除了点突变外,还有潜在的证据表明重组也是2019-nCoV进化的机制。
在比较全长S蛋白序列时,研究人员发现,2019-nCoV与人和蝙蝠SARS病毒的序列同源性为39%,与MERS或其他冠状病毒的同源性则为29%。
值得注意的是,研究团队发现2019-nCoV和穿山甲冠状病毒在S蛋白的RBD(氨基酸315-550区域)中共享几乎相同的氨基酸序列,但与RaTG13却不相同。
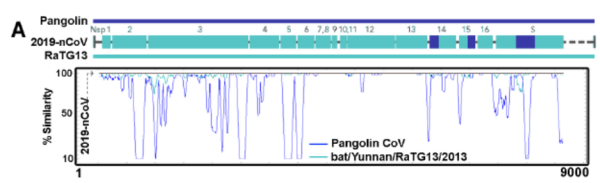
穿山甲CoV(蓝线)和RaTG13 / 2013(绿线)与2019-nCoV的氨基酸同源性对比图,保守受体结合结构域(RBD)以黄色突出显示
为了证实这一发现,研究团队将此前别的课题组发表的穿山甲CoV与先前分离但未发表的穿山甲CoV序列进行了比较。
基于比对和系统发育分析,研究人员发现2019-nCoV的共有序列与BetaCoV / 穿山甲/广东/P2S/2019(EPI_ISL_410544)具有最高的同一性,而在广西分离的穿山甲CoV株则发现存在其他突变和插入缺失。
接下来,研究人员使用ML方法(Maximum likelihood,最大似然法)检测了2019-nCoV的共有S蛋白序列与25种代表性CoV病毒株(包括Hu-CoV,SARS和MERS)和5新穿山甲CoV病毒株的系统发育关系。
结果表明,2019-nCoV的S蛋白可能从穿山甲冠状病毒衍生出来,而不是蝙蝠冠状病毒RaTG13,当然这两者都可能与蝙蝠-SARS-CoV或bat-SL-CoVZC542处于同一谱系。
他们提出,2019-nCoV的整个基因组结构与RaTG13最具有同源性,但S蛋白的RBD却与穿山甲CoV最具同源性,这一差异表明在2019- nCoV 的进化过程中,RaTG13样冠状病毒和穿山甲样冠状病毒病毒株之间可能发生了重组。
研究人员还进一步检查了基因组中的所有氨基酸突变。他们发现,当比较穿山甲样冠状病毒和2019-nCoV时,除了RBD外,nsp(非结构蛋白)14和15中的区域还共享连续序列(图3D)。
两个进化枝,以及独特的弗林蛋白酶切位点
为了进一步评估2019-nCoV与其他SARS冠状病毒的关系,研究人员分析了2019-nCoV和不同的人/蝙蝠SARS病毒的RBM(Receptor binding motif,受体结合基序),并观察到它们可以清楚地分为两个不同的进化枝。
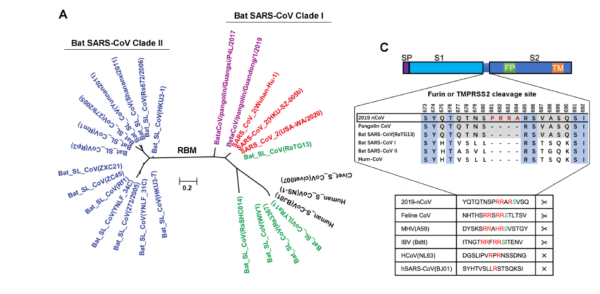
进化枝I病毒包括2019-nCoV、穿山甲CoV,以及12种蝙蝠SARS(蝙蝠SARS CoV I,例如RaTG13)和人类SARS病毒。
进化枝II病毒则包含了49种蝙蝠SARS病毒(bat SARS CoV II),例如ZXC21和ZC45,它们与2019-nCoV有大约90%的核苷酸和氨基酸同源性性。
上述两个进化枝之间的主要区别在于,进化枝II病毒的RBM存在5个、13-14个比进化枝I病毒短的氨基酸区域。
此前的研究表明,SARS病毒RBM的13-14个氨基酸区域形成独特的环结构,该结构通过两个半胱氨酸残基之间的二硫键稳定。尽管该环区中2019-nCoV的氨基酸序列与人SARS病毒的氨基酸序列非常不同,但两个半胱氨酸残基都是保守的。
有趣的是,所有已知使用ACE2作为进入受体的病毒都属于I型,而所有不使用ACE2进入受体的蝙蝠SARS病毒都属于II型。因此,研究团队预测,包括2019-nCoV在内的I型分支病毒可以通过人ACE2(血管紧张素转换酶2)感染宿主细胞,而II型分支病毒不能通过ACE2感染宿主细胞。
此前的研究得出的是,SARS病毒使用人ACE2作为受体来感染宿主细胞,而MERS病毒则是使用DPP4作为其受体。最近的一些表明ACE2也是2019-nCoV的进入受体,尽管其他宿主细胞因子如TMPRSS2也可能参与其中。
在进化枝II病毒中,尽管它们的总体基因组序列与2019-nCoV存在同源性,但尚无属于该枝的病毒利用ACE2进入受体的报道。
因此,这一研究不仅强调了RBM在确定进入受体特异性中的关键作用,而且提出了一个有趣的问题,即β冠状病毒的同源病毒株如何通过RBM中的插入、缺失或重组等突变来改变趋向性。
值得注意的是,研究团队还发现,2019-nCoV在S蛋白内或核苷酸位置23619-23632上有独特的四个氨基酸插入(681-PRRA-684)。
有趣的是,上述2019-nCoV的S蛋白中的这种插入(PRRA)为哺乳动物弗林蛋白酶蛋白创建了潜在的切割位点RRAR。
为了了解这种插入的独特性,研究团队使用了来自人、麝猫和蝙蝠的SARS-CoV株进行了序列比较。他们发现,这种插入是2019-nCoV特有的。当与其他冠状病毒家族成员进行比较时,研究人员发现也能够识别到类似的位于S蛋白的S1和S2域之间结构边界的插入。
而此前的研究则表明,病毒可能发生蛋白酶裂解,从而触发病毒-细胞膜之间的融合。这种启动和触发融合机制的灵活性极大地调节了不同冠状病毒的致病性和趋向性。
不过,此前在SARS-CoV中没有检测到这种蛋白酶裂解。在SARS-CoV中引入一个酶切位点,则会导致S蛋白的裂解并增强膜融合活性。此外,在SARS-CoV假型病毒中引入一个裂解的S蛋白,也会使其能够直接进入宿主细胞。
根据之前的测序和结构分析,2019-nCoV S蛋白被预测为可以与ACE2受体相互作用,触发与宿主细胞膜的融合并引发感染。因此,导致S1-S2亚基改变的突变或插入会显著影响病毒感染。
研究团队推测,PRRA的插入可能使S蛋白裂解,从而触发病毒融合事件。
虽然导致如此高感染率的确切机制仍有待进一步研究,但研究团队认为,他们的数据显示RBD重组、S1和S2域插入独特的弗林酶切点或TMPRSS2酶切位点,也许可以解释为什么这个新出现的病毒和其他SARS、MERS相关的β冠状病毒相比传染性显著增加。





- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




