- +1
Science封面:基因组基础模型 Evo,从分子到基因组理解生命复杂性
原创 董弘禹 集智俱乐部

导语
随着大语言模型时代的到来,各领域都涌现出了一批使用大数据、强算力训练出的基础模型,它们在评测中表现优异,并能泛化到各种下游任务。在 AI for Life Science 领域,单细胞基础模型 scGPT、scFoundation,蛋白大模型Alphafold3、ROSTTAFold 等相继涌现。2024年11月15日,美国 Arc 研究所(Arc Institute)和斯坦福大学的研究团队提出了一种基因组大模型 Evo,能够以无与伦比的准确性解码和设计从分子到基因组规模的 DNA、RNA 和蛋白质序列,打通“中心法则”,这一成果刊登在当期 Science 封面,为解码复杂生命系统提供了利器。
关键词:AI for Science,基因组基础模型,基因组设计
董弘禹 | 作者

论文题目:Sequence modeling and design from molecular to genome scale with Evo
论文链接:https://www.science.org/doi/10.1126/science.ado9336
在生活中,ChatGPT 可以写小说、编写计算机代码、提供出行建议,它能够阅读互联网上的所有语言文字信息,并生成问题的答案。在分子生物学中,DNA 序列是碳基生物的“语言文字”,读懂这些信息就能够掌握遗传密码。科学家已经开发了一些模型,可以像分析大语言模型中的单词一样分析 DNA 序列,如 DNABERT2 等。然而,这些模型只能解释和预测相对较短的 DNA 片段,并且训练数据也十分有限,泛化性能不高。基于此,科学家们研发了 Evo 模型。它以数十亿条基因序列为基础,可以推断出细菌和病毒基因组的运作方式,并利用这些信息设计新的蛋白质甚至整个微生物基因组。
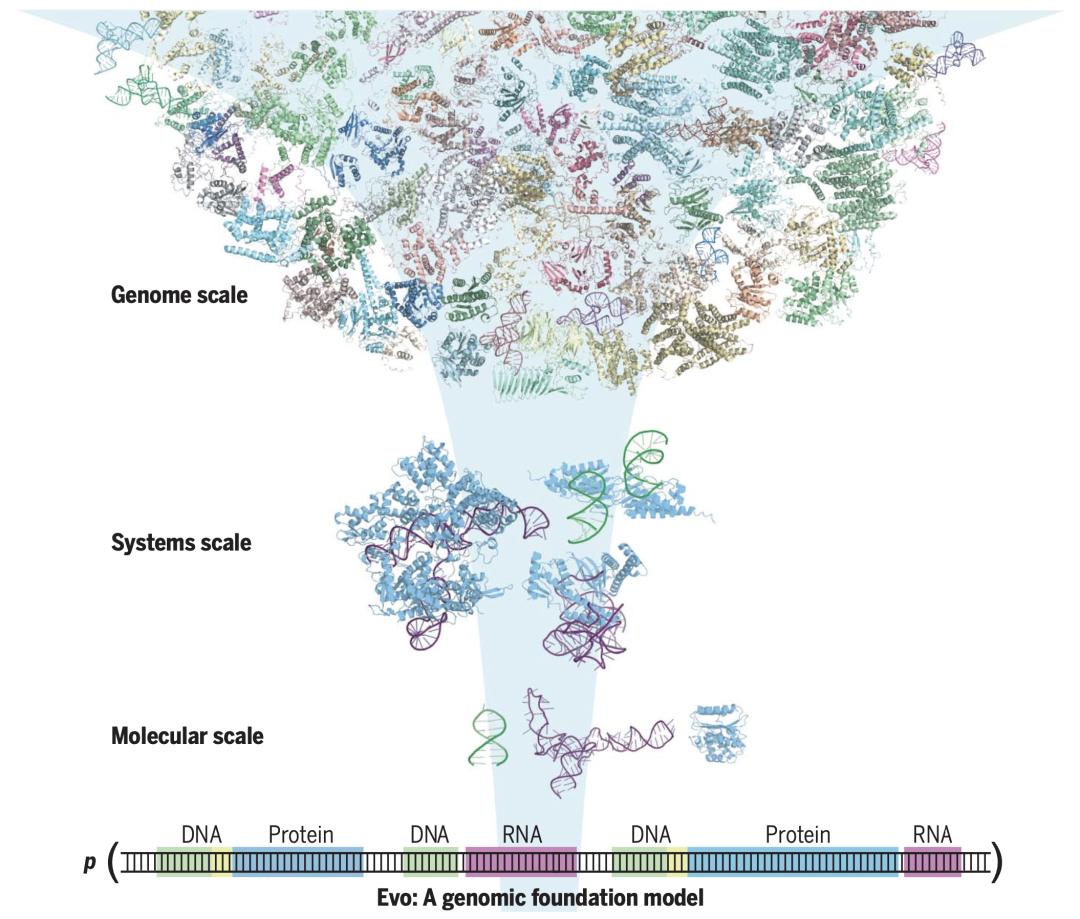
图1. 基因组基础模型 Evo 具有70亿个参数,可以学习从单个核苷酸到整个基因组的生命复杂性。
1. 基因组基础模型 Evo:架构与训练细节
想要理解大批量的基因数据,首先就要改进模型架构。Evo 采用了基于 StripedHyena 的框架,在 270 万个进化多样的原核生物和噬菌体基因组上进行预训练,从而获得对遗传语言的基本理解,预测 DNA 的功能或生成新的 DNA 序列。
StripedHyena 架构如图1B所示,该模型混合了密集二次 Transformer 算子和次二次型 Hyena 算子用于提高计算效率。同时,该模型将上下文窗口扩增到长达13万碱基,显著提高了模型识别基因与其他基因调控元件(如启动子、增强子等)之间联系的能力。为了为了确定 Evo 的最佳架构和缩放比例,图1F、G比较了在计算最优边界上预训练的不同模型的缩放率,在数据集大小和模型大小之间进行最佳计算分配。
从训练角度来看,为了防止恶意用户设计生物武器,研究人员从 AI 的训练集中删除了任何攻击人类或其他真核生物的病毒序列,并在接近 3000 亿核苷酸序列信息上进行了4周的训练。
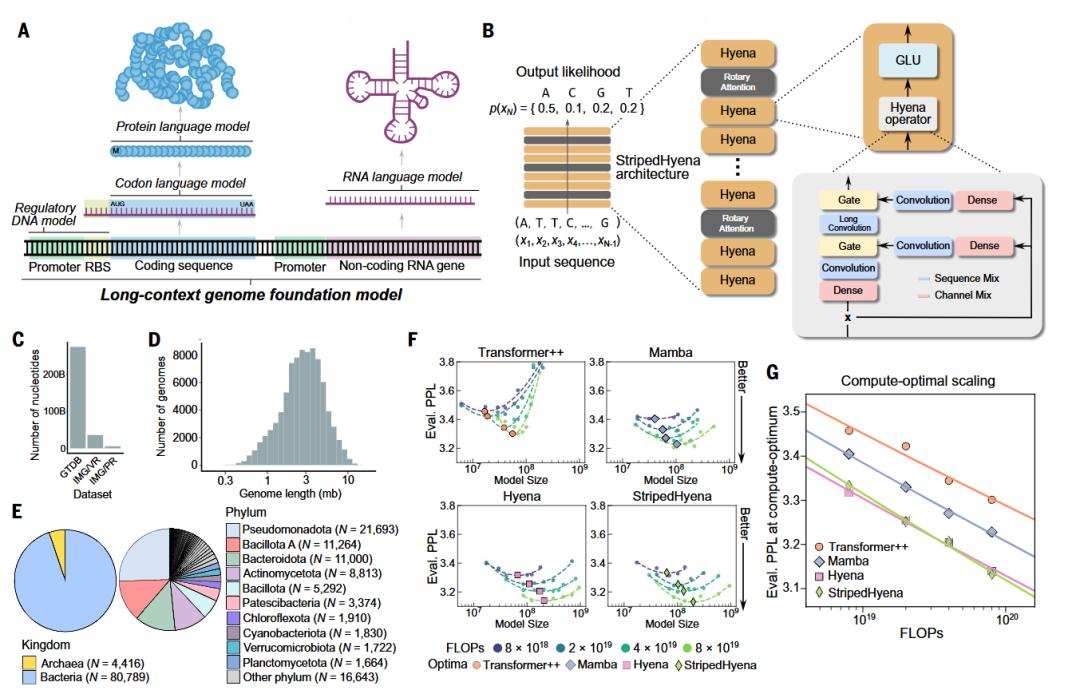
图2. 在原核生物中预训练基因组基础模型
2. Benchmark与性能比较
为了测试 Evo 模型的性能,研究人员首先衡量它是否能够预测突变对生物序列的影响。具体而言,在零样本功能预测的条件下使用 Evo 与其他模型,预测蛋白质突变对功能的影响、非编码 RNA 突变对功能的影响、调控 DNA 序列对基因表达的影响。图2展示其相关性的强度超过了之前从 DNA 序列数据推断突变效应的人工智能模型;其工作效果与其他依赖蛋白质序列的 AI 模型一样好。
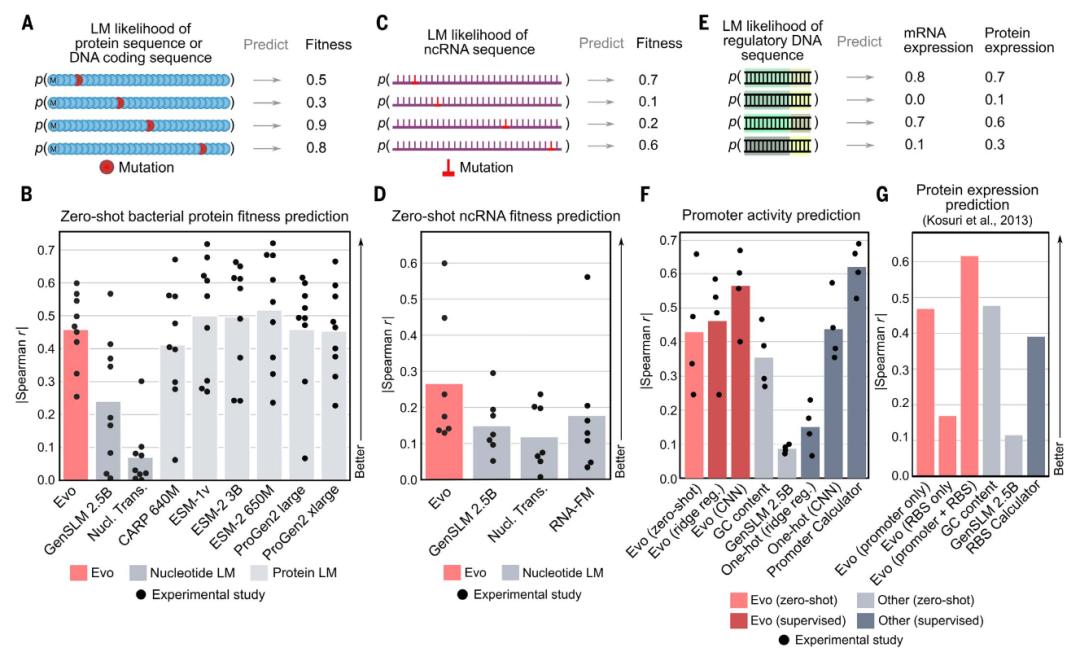
图3. Evo 对蛋白质、非编码 RNA 和调控 DNA 进行零样本功能预测
3. 下游应用:
从分子到基因组尺度的序列设计
除了判别式任务之外,基础模型也应有生成能力。ChatGPT 能够生成文章,Evo 模型也能够生成基因序列。为此,研究团队让 Evo 设计新版本的 CRISPR 基因编辑器。如图3所示,Evo 首先研究了 70,000 多个编码 Cas 蛋白及其伴侣 RNA 的细菌 DNA 序列。然后,该模型设计了数百万个分子的潜在版本。研究人员挑选了 11 个最有可能的 Cas9 变体,并在实验室中合成了这些蛋白质。在试管实验中,设计的 Cas9 酶中最好的一种,在切割 DNA 方面与商业版本的蛋白质一样好。
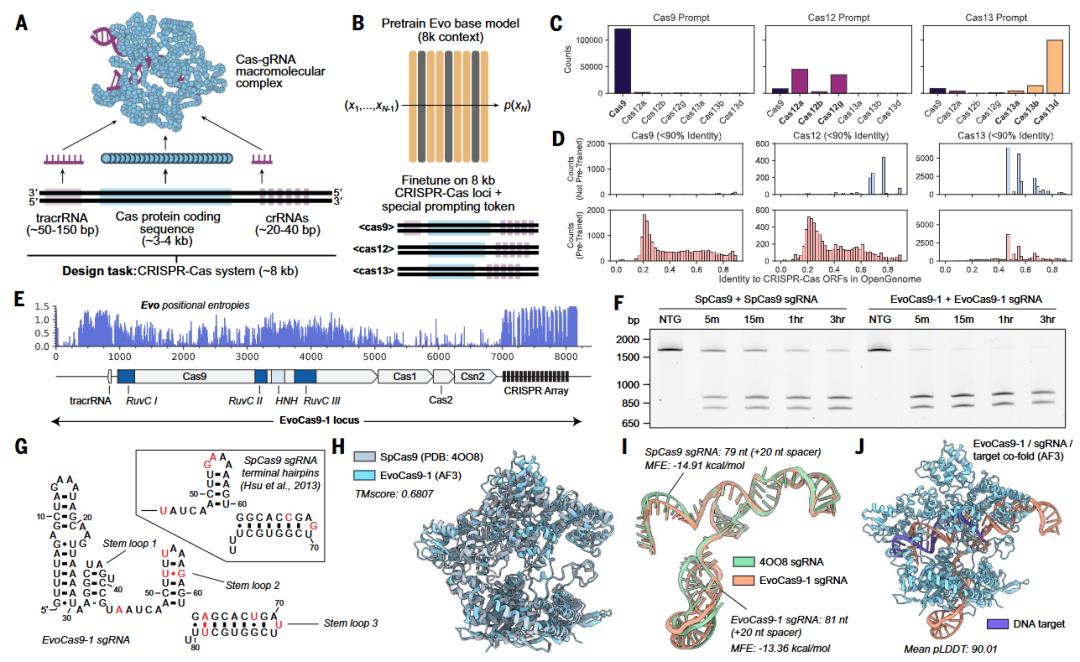
图4. 在 CRISPR-Cas 序列上进行微调可实现蛋白质-RNA 复合物的生成性设计
此外,Evo 还擅长多元件系统生成任务,如图4所示,团队通过对 CRISPR-Cas 序列和IS200/IS605 序列进行微调,可以实现合成 CRISPR-Cas 分子复合物和转座系统。研究人员实验验证了 Evo 生成的 CRISPR-Cas 分子复合物以及 IS200 和 IS605 转座系统的功能活性,这是使用语言模型进行蛋白质-RNA 和蛋白质-DNA 协同设计的第一个实例。
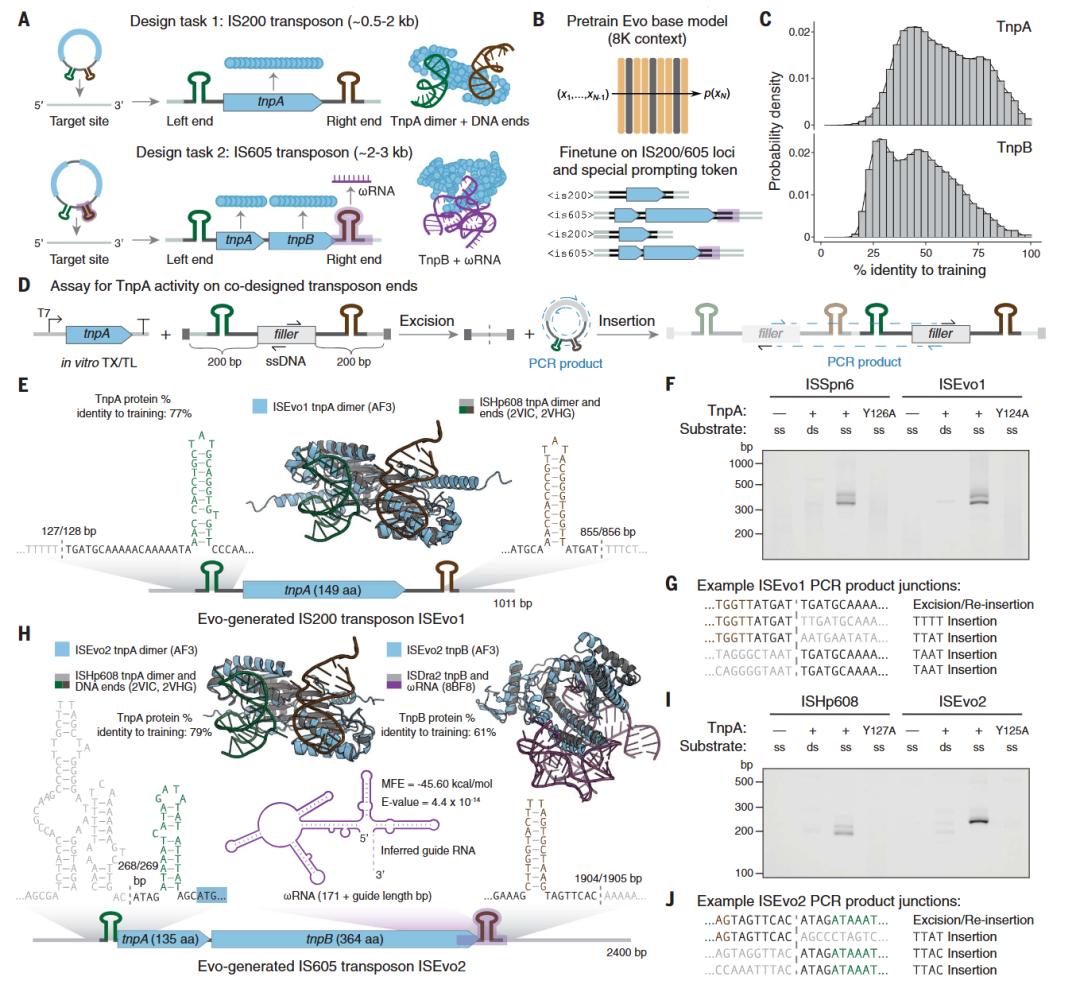
图5. 在 IS200/IS605 序列上进行微调可实现转座生物系统的生成性设计
最后,研究人员使用 Evo 生成了长达 1MB 的 DNA 序列作为细菌的基因组,这些序列展现出真实基因组的多个特征,包括编码密度、基因组织、密码子使用偏好性、四核苷酸使用模式等(图5)。
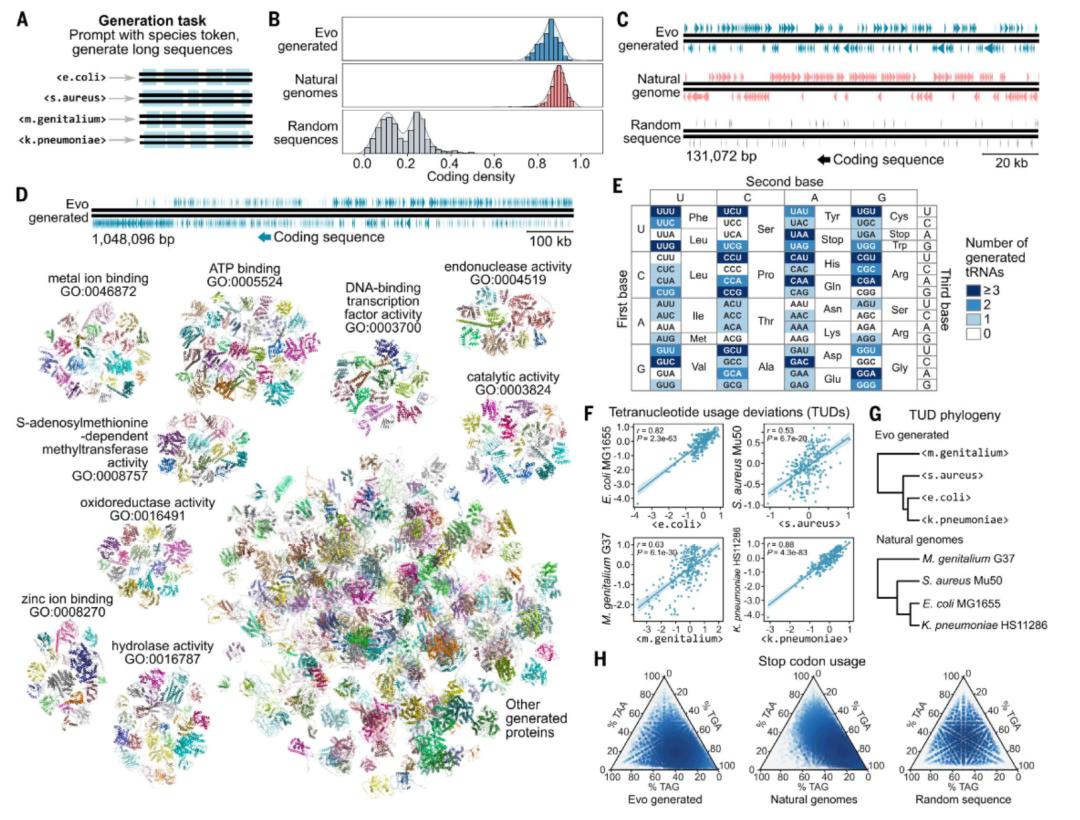
图6. Evo 生成具有密集编码架构的基因组规模序列
总体而言,Evo 经过 270 万个原核生物和噬菌体基因组的训练,展示了跨 DNA、RNA 和蛋白质模态的零样本函数预测,其性能可与特定领域的语言模型相媲美,甚至优于特定领域的语言模型。模型首次实现了单核苷酸分辨率下的长序列 DNA 建模,实现了从分子到基因组尺度的序列设计能力。这些突破为生物工程和基因组设计开辟了新的可能性。
原标题:《Science封面:基因组基础模型 Evo,从分子到基因组理解生命复杂性》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。





- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2025 上海东方报业有限公司




