- +1
Nature子刊:刘琦/张军/黄行许团队开发首个基因编辑AI大模型,从头识别新型基因编辑系统
编辑丨王多鱼
排版丨水成文
CRISPR-Cas系统是原核生物用来抵抗外来遗传元件(如噬菌体和质粒)的天然免疫机制,自2012年首次展现“基因剪刀”功能以来,已成为生命科学基础研究、农作物改良和基因治疗的关键技术。研究人员正在不断探索更高效、小型、靶向性强的新型基因编辑系统,以扩展其在基因编辑和治疗领域的创新应用。新系统的发现和开发为CRISPR技术开拓了更多可能性,加速了其在基础研究和临床转化中的应用。
在V型CRISPR系统的众多亚型中,存在以Cas12a为代表的一类特殊的Cas12同源蛋白。这些蛋白具备独立加工crRNA的能力,无需额外的RNA酶或tracrRNA,因此其生成的gRNA较短(约45nt),构成自加工pre-crRNA的CRISPR系统。类似的蛋白还包括Cas12i、Cas12h、Cas12j和Casλ。它们的共同特点是:1)具备RuvC D-E-D核酸酶活性位点,2)具备DNA切割和编辑能力,3)无需tracrRNA,4)具有自加工crRNA的能力。
这类Cas12同源蛋白在应用转化中潜力巨大,具有成为优质CRISPR工具的优势:1)sgRNA设计简单,2)sgRNA短且易于递送,3)支持多靶点基因编辑,4)避免tracrRNA相关的核心专利。从头识别该类同源蛋白,将有望形成新型有效的基因编辑工具,获得独立知识产权,突破基因编辑工具的知识产权垄断。
发现和筛选CRISPR系统需要准确识别其附近的Cas蛋白,特别是效应蛋白。主要有两种策略用于识别Cas蛋白:1)基于氨基酸序列相似性的方法,这种方法简单直接,如NCBI的PGAP和CRISPRCasTyper等生物信息学工具,依赖于BLAST比对和已知Cas蛋白的HMM模型搜索。然而,这种方法可能遗漏远缘同源蛋白;2)基于结构相似性的方法,这种策略基于生物学中“结构-功能”关系,即蛋白质的功能由其结构决定。随着蛋白质结构预测技术的发展,如AlphaFold和RoseTTAfold,结构预测在CRISPR-Cas的发现和功能筛选中发挥了重要作用。
然而,这些技术通常依赖多序列比对(MSA),而在Cas蛋白识别中,特别是某些V型和VI型亚型的效应蛋白,已知同源蛋白数量有限,限制了参考数据集的构建,显著增加了搜索的难度。因此,进行CRISPR-Cas系统的系统性识别和功能筛选在同源物有限和缺乏真实结构信息的情况下极具挑战。
2024年11月19日,同济大学生命科学与技术学院生物信息学系、同济大学-上海自主智能无人系统科学中心刘琦教授团队联合南京医科大学生殖医学与子代健康全国重点实验室张军教授团队和浙江大学医学院附属第一医院黄行许教授团队,在 Nature Communications 期刊发表了题为:Discovering CRISPR-Cas system with self-processing pre-crRNA capability by foundation models 的研究论文。
该研究开发了领域内首个可用于识别具备自加工功能的Cas12蛋白的蛋白质序列AI大模型CHOOSER(Cas Homlog Observing and Self-processing screening),基于蛋白质语言模型进行Cas酶的挖掘及pre-crRNA自加工功能的预测。
借助CHOOSER,研究团队发现了11种新的Casλ同源蛋白,几乎将该亚型的已知数量翻倍。实验验证表明,其中一种新发现的EphcCasλ蛋白具备自我处理pre-crRNA、DNA切割及转切割的能力,展现出其在基因编辑和CRISPR病原体检测中的应用潜力。

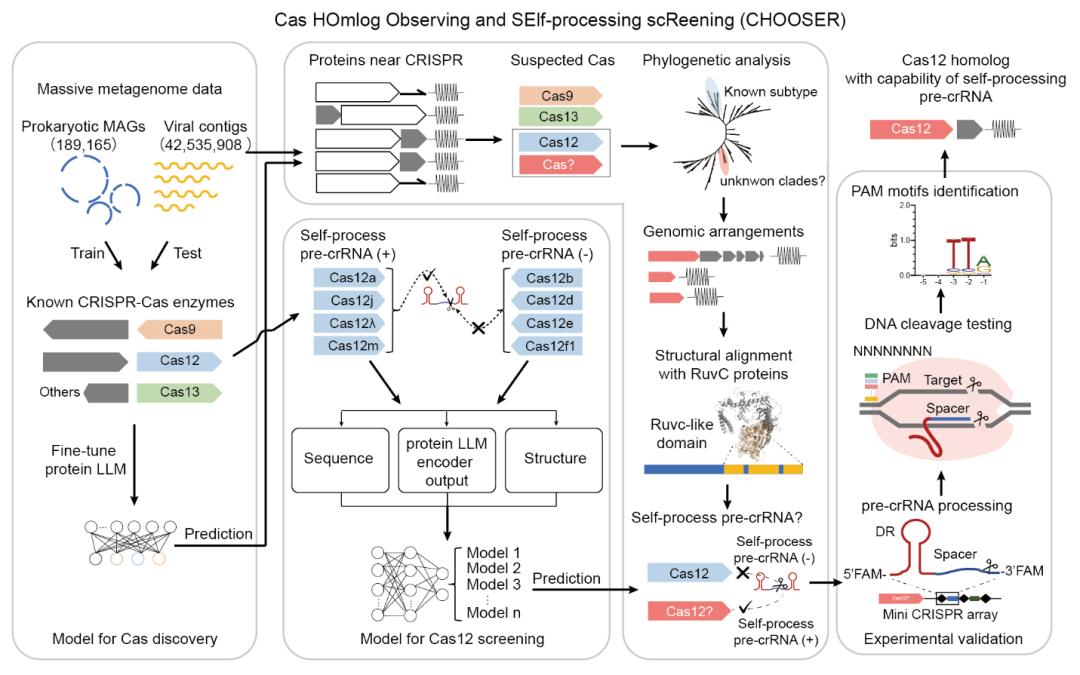
图1. CHOOSER计算框架示意图
CHOOSER旨在解决CRISPR-Cas系统识别和功能筛选中的两大关键问题:
1)运用AI大模型策略,通过微调预训练的大型语言模型ESM-2来发现远缘的Cas同源物;
2)利用基础模型生成的表征,直接预测Cas12酶的特定功能,在本应用中即自我处理pre-crRNA的功能。
具体来说,CHOOSER成功识别了3477个潜在的CRISPR-Cas系统,扩展了已知的II型、V型和VI型系统的数量。CHOOSER在这些系统中发现了39个此前未被现有基于比对的工具(如CRISPRCasTyper)识别的Cas12候选者,并在其中鉴定出11个Casλ同源物,所有这些同源物均被预测为具备自我处理pre-crRNA的功能。实验验证进一步确认了其中一种名为EphcCasλ的Casλ同源物的pre-crRNA处理活性和DNase活性。
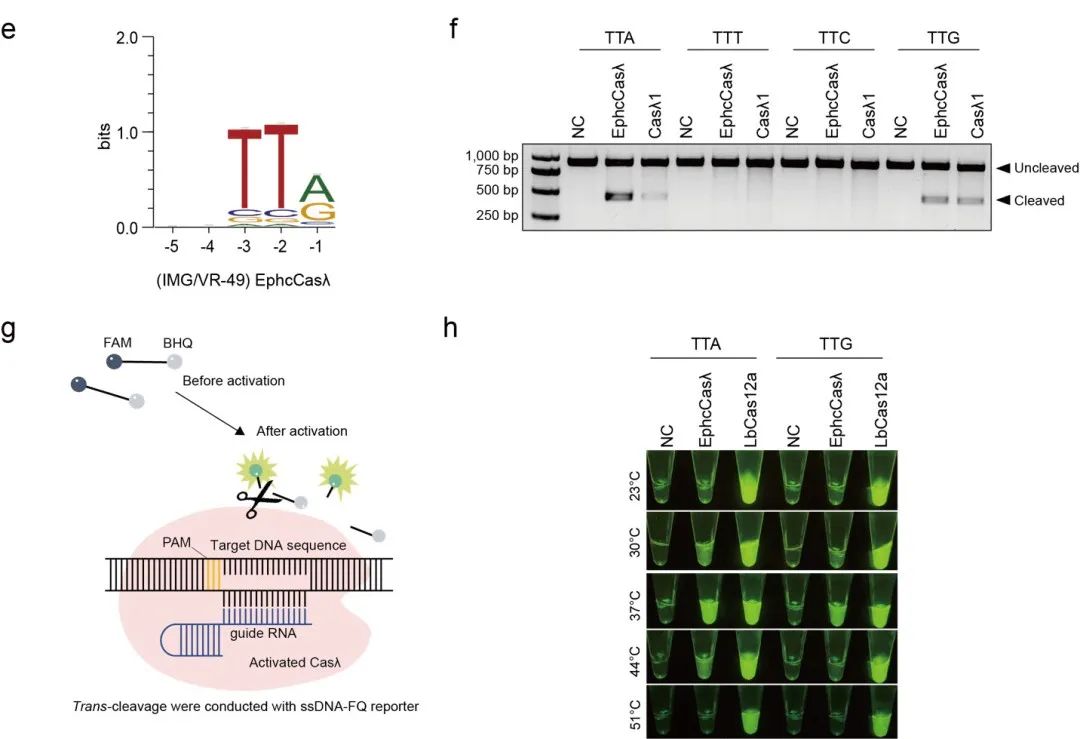
图3. EphcCasλ的pre-crRNA处理活性和DNase活性验证
总结来说,该项工作表明在缺乏真实结构信息的情况下,蛋白质LLM生成的表征可用于识别和筛选具有有限标记的Cas同源物的CRISPR-Cas系统。CHOOSER大模型的计算分析和实验验证提供了通过基础模型(Foundation Model)发现具有特定功能的CRISPR-Cas系统的创新策略,形成了一种创新的基于基础模型的CRISPR-Cas系统功能发现的普适AI框架,未来将有望进一步拓展其在基因编辑de-novo识别中的广泛应用。
该AI大模型由同济大学刘琦教授团队、南京医科大学生殖医学与子代健康全国重点实验室张军教授团队和浙江大学医学院附属第一医院黄行许教授团队联合开发。同济大学李文慧和之江实验室姜先月为该论文共同第一作者。
论文链接:
https://www.nature.com/articles/s41467-024-54365-0
原标题:《Nature子刊:刘琦/张军/黄行许团队开发首个基因编辑AI大模型,从头识别新型基因编辑系统》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。





- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




