- +1
通过超图学习梳理基因组规模代谢网络的缺失反应|Nat. Commun. 速递
原创 集智编辑部 集智俱乐部

关键词:代谢网络,深度学习,超图学习

论文题目:Teasing out missing reactions in genome-scale metabolic networks through hypergraph learning
论文来源:Nature Communications
论文链接:https://www.nature.com/articles/s41467-023-38110-7
基因组规模代谢模型(GEMs,GEnome-scale Metabolic models)是预测生物体细胞代谢和生理状态的有力工具。它通过两个矩阵提供了全面的基因-反应-代谢关联,是有机体代谢的数学表示。然而,由于人们对代谢过程的知识并不完备,即使是精心设计的 GEMs 也存在知识空白(例如,一些缺失反应)。现有的空白填补方法通常需要表型数据作为输入,我们仍然缺乏一种快速准确的计算方法,在获得实验数据之前,填补代谢网络的空白。
在这篇最近发表在 Nature Communications 的文章中,作者提出了一种基于深度学习的方法——切比雪夫光谱超链接预测器(CHESHIRE,CHEbyshev Spectral HyperlInk pREdictor)——仅仅从代谢网络的拓扑性质来预测 GEMs 中的缺失反应。作者用 CHESHIRE 预测了926个高质量和中等质量的 GEMs 里人工去除的反应,证明了 CHESHIRE 优于其他基于拓扑的方法。此外,对发酵产物和氨基酸分泌物的49个草拟 GEMs,CHESHIRE 能够提升表型预测。这两种类型的验证表明,CHESHIRE 是 GEM 管理的强大工具,可以揭示反应和观察到的代谢表型之间的未知联系。
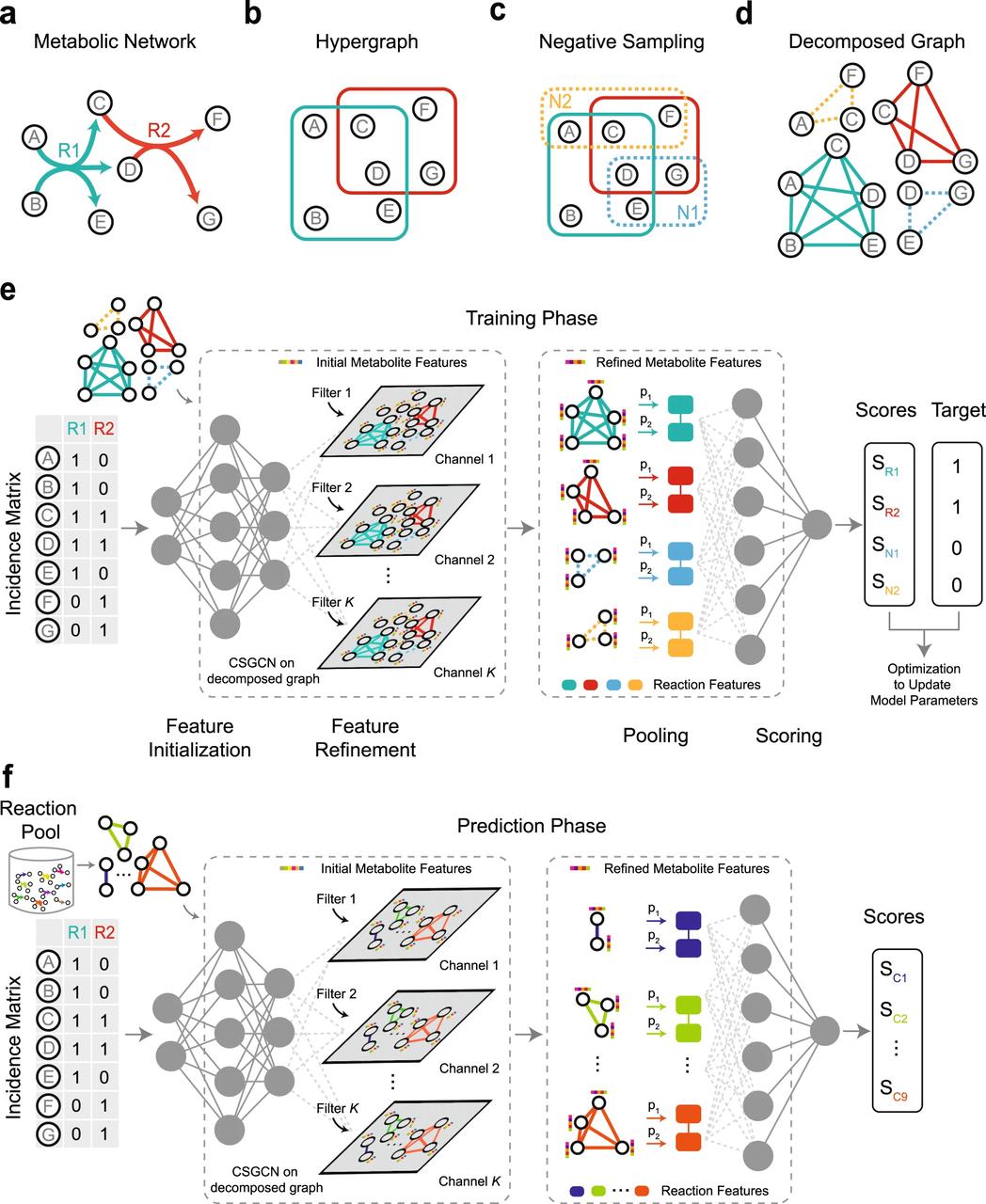
图1. CHESHIRE 工作流程。
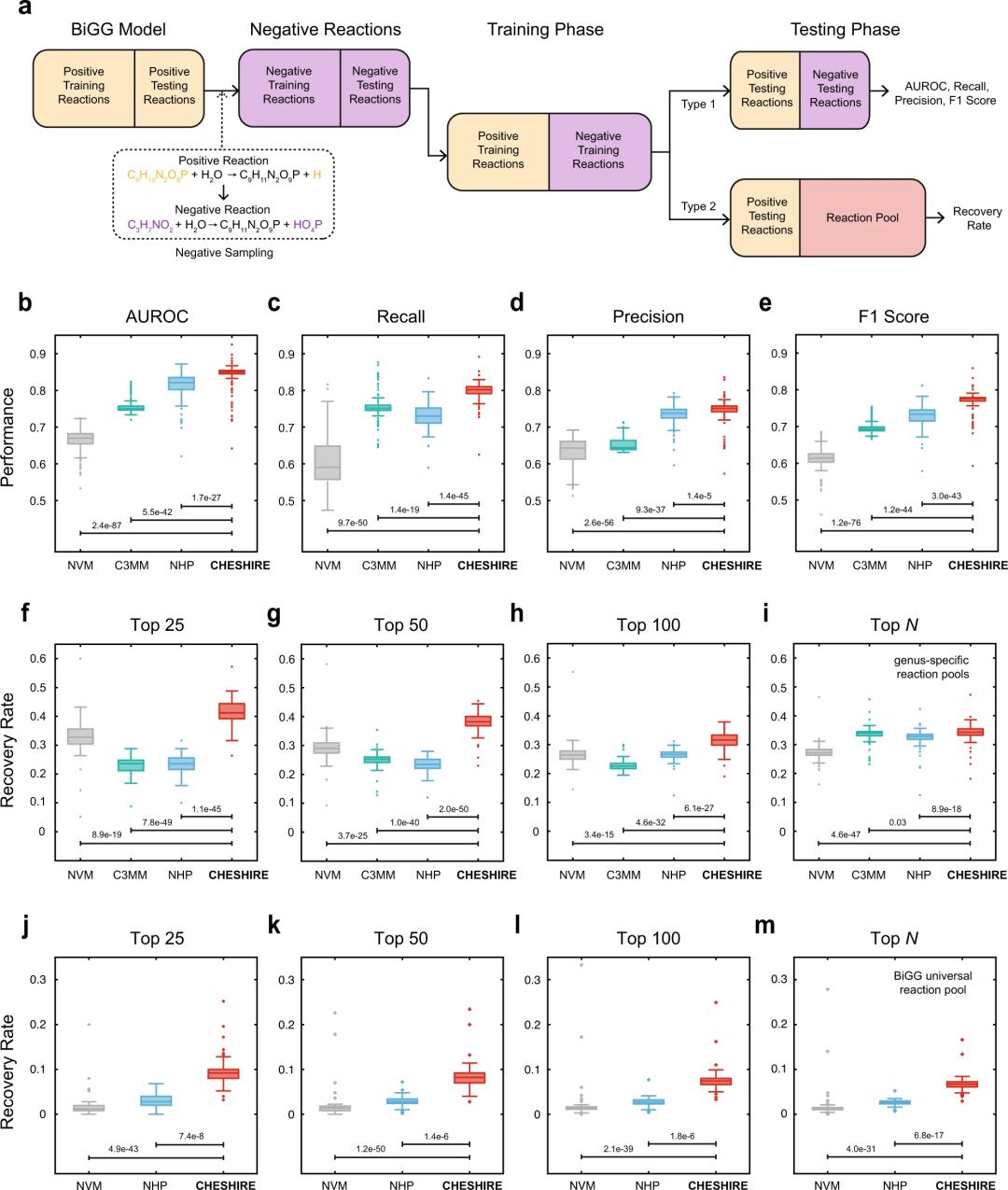
图2. 人为引入缺失反应,进行内部验证。
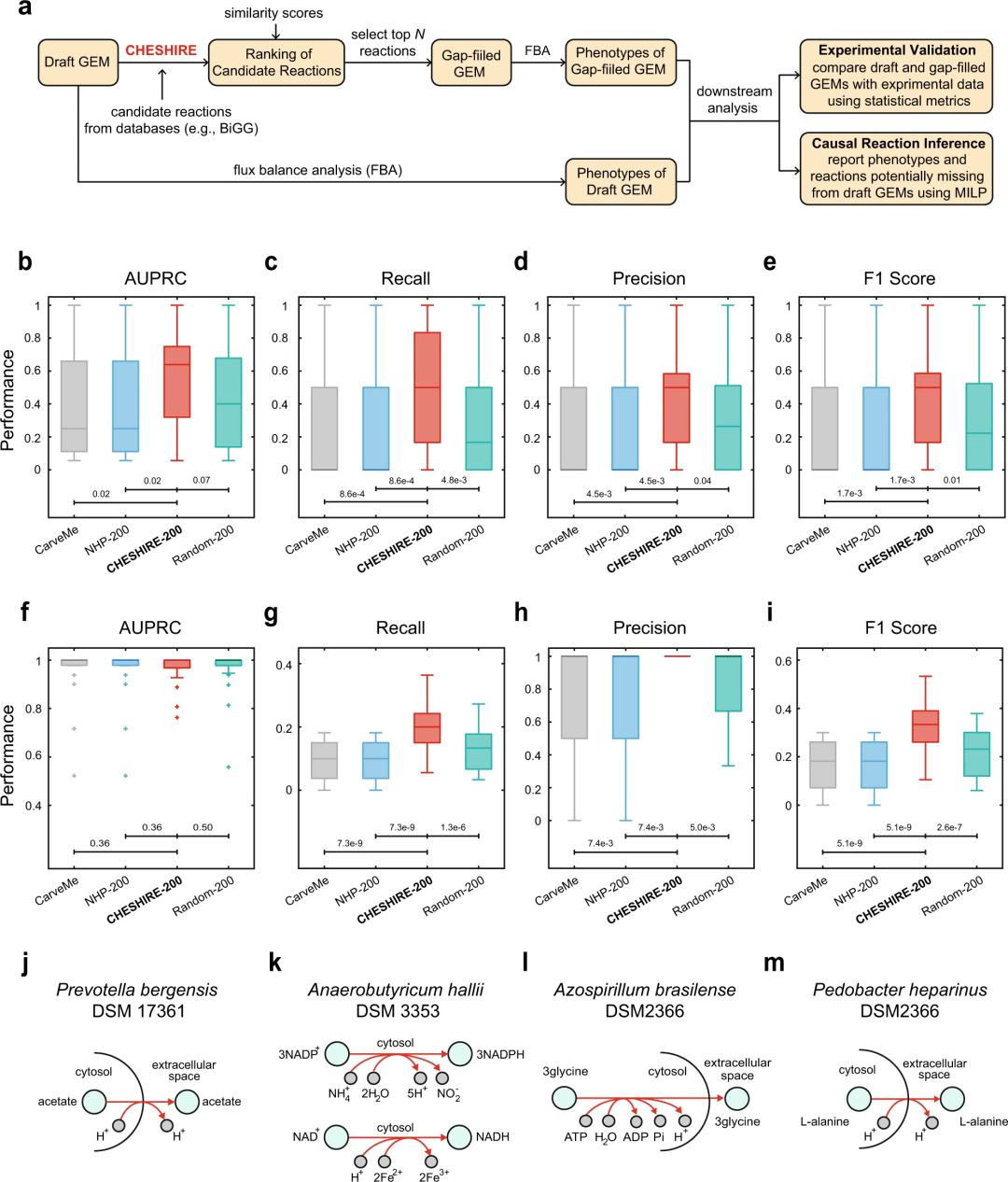
图3. 通过预测代谢表型进行外部验证。
编译|汪显意
原标题:《通过超图学习梳理基因组规模代谢网络的缺失反应|Nat. Commun. 速递》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。





- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2025 上海东方报业有限公司




