- +1
【科技前沿】Mol Cell | 薛愿超团队开发CRIC-seq技术揭示PTBP1介导RNA成环调…
在真核生物中,同一个pre-mRNA可通过可变剪接产生不同的mRNA剪接异构体,进而表达功能相似或不同的蛋白质。可变剪接的异常会导致癌症、神经退行性疾病和心脑血管疾病等重大疾病的发生。在可变剪接过程中,RNA结合蛋白(RNA-binding protein, RBP)通常结合在pre-mRNA的不同位置,以促进或者抑制可变外显子的使用,这种调控机制被称为位置效应。目前,由于技术手段的缺乏,位置效应产生的深层次分子机制仍不清楚。
2023年3月22日,中国科学院生物物理研究所薛愿超课题组在Molecular Cell杂志在线发表了题为 Capture RIC-seq reveals positional rules of PTBP1-associated RNA loops in splicing regulation 的研究论文,创建了一种捕获特定RBP介导的RNA-RNA原位空间互作的新技术CRIC-seq(Capture RIC-seq)。利用该技术,研究人员绘制了PTBP1、hnRNPA1和SRSF1介导的RNA空间互作图谱,揭示了PTBP1通过二聚化介导两侧内含子之间形成跨越可变外显子的RNA环从而抑制剪接,以及介导一侧内含子内部形成RNA环以促进可变外显子剪接的新机制。此外,该研究发现癌症相关剪接数量性状位点 (splicing quantitative trait loci, sQTLs) 可破坏PTBP1蛋白与RNA靶位点的亲和力、影响RNA环的形成,进而导致剪接异构体变化和癌细胞增殖异常。

CRIC-seq的基本流程是对细胞进行甲醛交联以固定RBP介导的RNA-RNA空间互作,经过细胞透化和MNase酶片段化处理之后,在RNA片段的3’末端标记pCp-Biotin,随后利用T4 RNA连接酶将空间邻近的RNA片段连接起来形成嵌合体RNA片段,然后利用高特异性的抗体富集特定RBP所结合的RNA片段,并进一步富集带有生物素标记的嵌合体RNA片段,以构建链特异性文库用于高通量测序(图1A)。此外,在CRIC-seq文库的构建过程中,研究者平行构建了IgG IP组以及不加pCp-biotin标记组(-pCp组)作为对照文库,以实现对实验组中背景信号的扣除(图1B-C)。
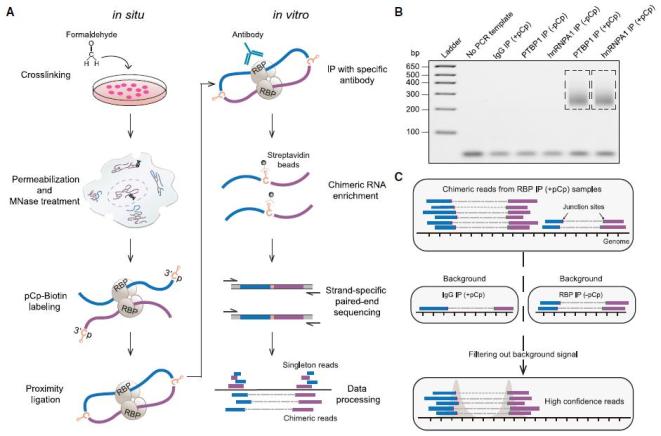
图1. CRIC-seq可准确捕获RBP介导的RNA-RNA空间互作。
随后,研究者分别分析了PTBP1、hnRNPA1和SRSF1介导的RNA环(远距离的RNA-RNA片段在蛋白质的介导下形成空间临近互作)在其调控的可变外显子上下游的分布模式。与hnRNPA1类似,但是与SRSF1不同,研究人员发现:在促进的可变外显子中,PTBP1介导的RNA环倾向于富集在某一侧内含子的内部;而在抑制的可变外显子中,PTBP1介导RNA环则倾向于跨越可变外显子并在两侧的内含子之间形成(图2)。上述研究结果表明,PTBP1能够以位置依赖的方式介导形成特异的RNA环并参与pre-mRNA可变剪接调控过程。
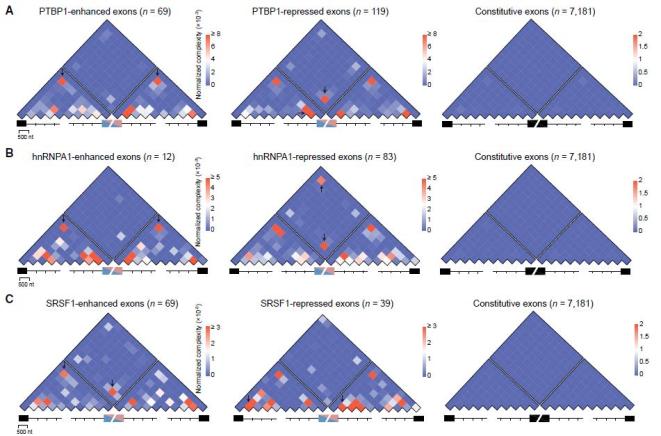
图2. PTBP1介导RNA成环调控可变剪接的规律。
PTBP1能够借助第23位半胱氨酸(Cys23,C23)形成同源二聚物,且不依赖于RNA或者DNA分子(图3A)。据此,研究者利用shRNA在HeLa细胞中敲降PTBP1,并在此细胞中分别表达野生型PTBP1或不能形成二聚体的突变型PTBP1(第23位半胱氨酸突变为丝氨酸,记为C23S)。检测发现只有表达野生型PTBP1才能回补PTBP1敲降后可变外显子的剪接变化,而表达C23S突变型PTBP1则不能回补可变外显子的剪接变化。这一结果表明PTBP1的二聚化能力对于其发挥可变剪接调控作用是至关重要的。
为了探究一些非编码区突变是否影响PTBP1调控剪接的过程进而影响肿瘤发生发展,研究者整合了CancerSplicingQTL数据库中的在癌症样本中与剪接存在统计关联的sQTL(splicing quantitative trait loci)与RNA空间互作信息,共发现121个可能会影响PTBP1介导RNA空间互作的sQTL位点。进一步,利用剪接报告基因实验,研究者发现sQTL位点会导致CHTOP和HERPUD2中可变外显子的异常剪接。更重要的是,HERPUD2内含子中的sQTL位点会促进增强HeLa细胞增殖能力的短转录本的产生。这展示了癌症相关的非编码突变有可能通过改变特定蛋白质介导的RNA空间互作调控可变剪接,进而影响肿瘤细胞的增殖。
综上所述,该工作建立了能够广泛用于捕获特定RBP介导的RNA空间互作信息的CRIC-seq新技术;揭示了PTBP1同源二聚体介导RNA 空间互作调控可变剪接的位置效应机制,从RNA 空间相互作用角度解释了部分非编码区突变的致病原因,为疾病关联非编码区突变的致病机理研究提供了新的思路和角度。
生物物理研究所博士研究生叶融和呼乃婧是该论文的共同第一作者,薛愿超研究员是通讯作者。此外,温州医科大学附属眼视光医院的硕士研究生许诗晗和杨晨作为共同作者参与了该课题的部分研究工作,并得到了周翔天教授的指导。
原文链接:https://do-i.org/10.1016/j.molcel.2023.03.001
本文转载自公众号“BioArt”
中国生物物理学会官方订阅号,为BSC会员及生物物理领域专业人士服务。
投稿及授权请联系:bscoffice@bsc.org.cn。
原标题:《【科技前沿】Mol Cell | 薛愿超团队开发CRIC-seq技术揭示PTBP1介导RNA成环调控可变剪接的新机制》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。




- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




