- +1
《细胞》:上海科技大学池天课题组发明新型基因打靶技术,绘制世界首张“扰动图谱”
*仅供医学专业人士阅读参考


在哺乳动物体内,存在着大约2万个用于编码蛋白的基因以及至少500种不同的细胞,因此至少有1000万种基因-细胞组合。
人类基因组序列于2001年已基本测毕,但在多数情况下,特定基因在特定细胞的功能至今依然迷雾笼罩,这已构成人类疾病诊断、治疗的一个重要瓶颈[1, 2]。
如果能系统性地将每个基因分别在每种细胞内敲除并鉴定其直接表型(存活、转录组、蛋白组等方面),从而描绘出“扰动图谱”(Perturb-Atlas),这将成为生物医学研究的一个分水岭。此后,探索基因的基本功能将像现在查找模式生物基因序列一样,只需简单地点击鼠标,这将大大加速生物医学研究。
近日,上海科技大学池天课题组在《细胞》发表重要研究成果,他们报道了一种基因打靶技术——iMAP(inducible Mosaic Animal for Perturbation),可通过药物诱导,在小鼠全身敲除100个基因,但在每个细胞只能随机敲除其中之一,从而将小鼠转化为嵌合体动物[3]。
这种新型动物可用于鉴定100个靶基因在全身各种细胞中的功能,同时也能通过简单的配繁衍生出100个传统的单基因敲除品系以用于后续验证等实验。
作为概念验证研究,池天课题组检查了90个基因敲除后分别对39种器官/组织/细胞(后文统一称“组织”)丰度的影响,描绘了世界第一张“扰动图谱”。
虽然这张图谱只是full-size图谱的雏形,但已提供了大量难以用其他技术轻易获得的宝贵信息,揭示了iMAP在基因组解码中的巨大潜力。上海科技大学的刘波、荆征宇、张校铭、陈玉鑫、毛少帅作为论文的共同第一作者。

论文首页截图
传统的小鼠单基因打靶技术不适合描绘扰动图谱,这不但是由于其通量低,还因为这种敲除常会导致严重的继发病变从而混淆表型。
基于pooled sgRNA病毒文库的“小鼠体内CRISPR基因筛选”,能够克服这些局限性[4, 5]。这是因为这种筛选不但通量高,而且由于文库中的任何一种sgRNA只能影响靶细胞群的极少部分成员(“嵌合型扰动”),所以通常不会破坏组织的整体功能而造成严重继发病变。
但是,体内CRISPR筛选亦有其短板:其主要形式为原位筛选(将病毒注入小鼠以感染靶细胞)[6-9]和移植筛选(先取出靶细胞,体外感染后回输体内)[10-14]。前者只适用于实质性器官,同时病毒感染效率低下且不均;后者则剥夺了靶细胞的空间信息,同时只适合少数既可移植又可感染的细胞类型(主要是血细胞)。
其次,无论是原位还是移植筛选,其试剂(病毒文库及取出的靶细胞)宝贵且不能重复使用,需反复制备才能完成扰动图谱这一庞大项目,从而使实验价格昂贵、操作复杂并遭受“批次效应”的干扰。

池天课题组开发的iMAP技术具有病毒文库的优点(高通量、嵌合型扰动),却无其短板(sgRNA遍布全身,小鼠品系能重复使用),从而使原位筛选变得简单易行,更使扰动图谱变得可望而可及。
接下来,我们就一起来看看iMAP技术究竟为何这么厉害。
iMAP融合了CRISPR-Cas9和Cre-Lox技术,其核心是由多达100个sgRNA表达单位串联而成的新型转基因,每单位两侧都安装了LoxP,整个序列被置于U6启动子下游(图 1A)。
转基因通常只表达第一个sgRNA,而利用他莫昔芬激活Cre,可使转基因发生重组,其余的sgRNA从而得以全部表达,但每个细胞只随机表达其中一个(图1B)。这些sgRNA可在Cas9的帮助下敲除相应的靶基因,从而将小鼠转化为嵌合体(图1C)。
最后得到的嵌合体可直接用于表型鉴定(包括描绘扰动图谱、原位筛选),也可衍生出上百基因单敲品系(图1D)。值得一提的是,iMAP品系一旦制成,是可反复利用的永久资源。
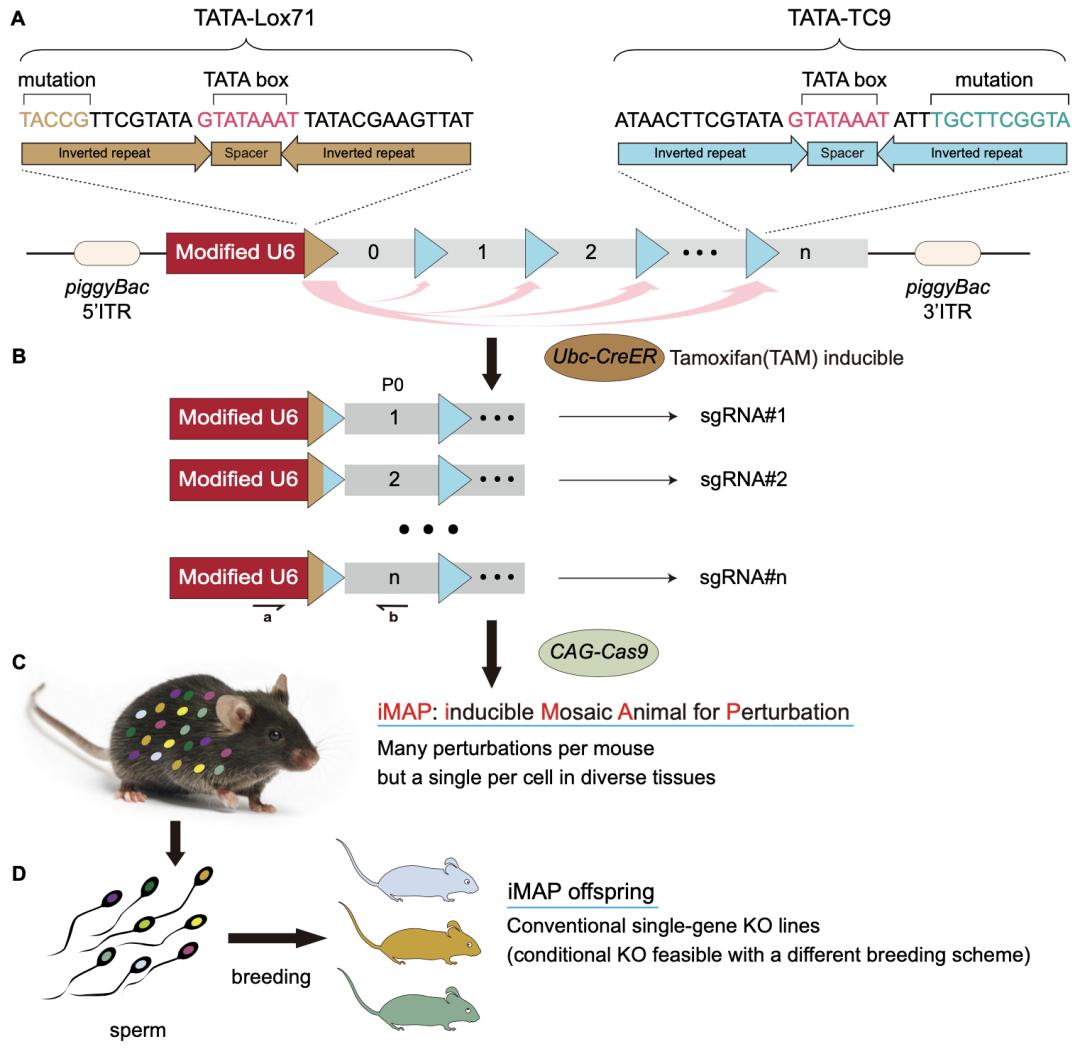
图1. iMAP原理。
(A)重组前的转基因。图示2个LoxP突变体TATA-Lox71和TATA-TC9,前者镶嵌于U6启动子中。只有U6启动子紧下游(位于P0位置)的sgRNA能表达。
(B)重组后的转基因。Ubc-CreER广泛表达CreER融合蛋白,后者被他莫昔芬激活,催化重组,使转基因上其他sgRNA随机前移到P0位置而得以表达。TATA-Lox71 和TATA-TC9重组产生的杂合LoxP不能继续重组,因此,每个细胞始终只能表达一个sgRNA.
(C)嵌合体鼠。CAG-Cas9广泛表达Cas9.
(D)嵌合体雄鼠能繁育生出多个单基因敲除品系。
为验证上述策略的可行性,研究人员选取了三类功能已知的基因,即细胞必需基因、抑癌基因和细胞表面受体基因,其敲除应分别导致细胞丢失、细胞扩增和细胞标记物丢失。
每类基因选取了两个基因,针对每个基因设计10个sgRNA(共60个),将其串联成“61-guide”转基因,发现其至少能稳定地传13代。在携带61-guide转基因并广泛表达CreER和Cas9的“iMAP-61”品系里,他莫昔芬能使转基因重组,释放各种sgRNA。
为鉴定sgRNA的生理效应,研究人员检测了全身10种组织的表型,发现其与预期的基本一致(例如敲除抑癌基因能使某些组织明显扩增乃至发生肿瘤),表明了iMAP的可行性。
另外,靶向同一基因的10个sgRNA,平均9个可测到活性,尽管这些sgRNA未经事先验证,而较明显的脱靶效应仅见于一个数据点(靶向必需基因的一个sgRNA在睾丸中被富集2.3倍)。这些数据不但表明iMAP确实可行,并且提示用一个(而不是多个)sgRNA靶向一个基因将是最佳设计,因为它使iMAP通量最大化但不明显增加假阳/阴性事件。
值得注意的是,靶向必需基因的sgRNA,其丰度在各组织中普遍降低,但其下降幅度有组织特异性,更新快的组织表现出了更大的下降幅度,提示细胞分裂能促进Cas9切割而提高对sgRNA的敏感性。
研究人员继而转向功能未知的基因。他们聚焦“胚胎必需基因”(占小鼠基因30 %),其敲除将导致胚胎死亡,因此其成体功能大多并不清楚。
作者挑选了87个在成体内广泛表达的胚胎必需基因,制备了新品系iMAP-100,它共携带100个sgRNA,其中87个靶向上述87个胚胎必需基因,3个靶向非必需对照基因,8个是non-targeting control。
用他莫昔芬诱导幼年鼠,3-4个月后检查,发现所有雄鼠睾丸萎缩,部分小鼠出现多囊肾(图2A)。同时,检测上述98个sgRNA在39种组织中的丰度,获得3822个组织-sgRNA的组合(数据点),构成了扰动图谱(图2B)。
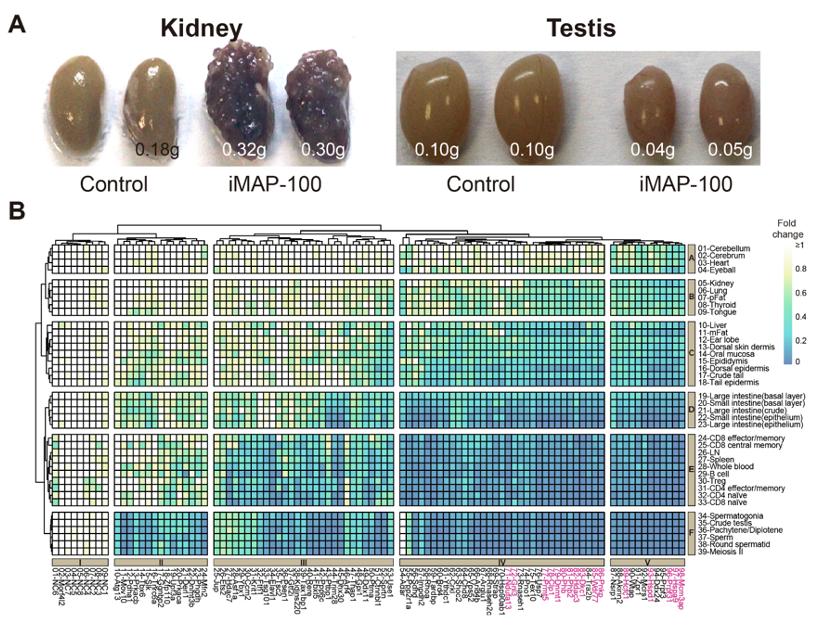
图2. iMAP-100表型。
(A)多囊肾和睾丸萎缩
(B)扰动图谱。展示98个sgRNA在39种组织中的丰度。为清晰展示丰度的下降,本图的color scheme将丰度上升当成丰度不变。人类CEG的同源基因(粉红色)富集于Cluster IV-V, 提示这些基因一般也为小鼠多种乃至全部组织所必需。
池天课题组的主要发现包括:
一、有61%的数据点,其sgRNA丰度明显变化,其中升高和减少的数据点分别占1%和60%,后者涉及所有(87个)胚胎必需基因,表明这些基因在成体内都有重要功能。其余39%的数据点,其sgRNA无明显变化,其中包括所有非靶向sgRNA和一个非必需基因的sgRNA(其余2个非必需基因的sgRNA仅有轻度改变),提示sgRNA无明显脱靶效应。
二、上述丰度升高的sgRNA,最典型的是靶向Tsc2的sgRNA,它在多囊肾中被富集95倍,提示Tsc2敲除能导致多囊肾,这与文献报道吻合。丰度下降的sgRNA分两类。
第一类由12个sgRNA组成,在所有(39种)组织都减少,提示其靶基因调控细胞的基本生理过程(图2B,Cluster V)。这种基因称Core Essential Gene(CEG)在合成生物学和肿瘤研究领域有重要意义。一般需在多种肿瘤品系中进行CRISPR筛选才能系统性地发现CEG,但iMAP则使生理条件下的系统性发现成为可能。
丰度下降的第二类sgRNA,其作用呈现组织特异性,其中有些靶点具有潜在临床价值(Cluster II-IV)。例如,Mov10 sgRNA只在男性生殖细胞减少,提示Mov10抑制剂是安全的男性避孕药;Derl1 sgRNA的减少只见于B细胞和男性生殖细胞,提示Derl1抑制剂是相对安全的抗肿瘤药物(DERL1是致癌基因)。
最有趣的是,Hdac7 sgRNA在初始CD8 T细胞严重减少,但在效应/记忆CD8 T细胞中明显恢复,提示Hdac7的敲除能促进CD8 T细胞的激活。事实证明,敲除人类CAR-T细胞的HDAC7的确能够促进杀瘤。
三、对90个targeting sgRNA,不同组织有不同敏感性,这取决于细胞分裂和其他细胞特征。在39种组织中,对sgRNA最敏感的是精原细胞,因为这种细胞里,有99%(89/90)的targeting sgRNA发生了改变,其中多为严重减少。这解释了睾丸独特的表型(萎缩),也提示精子发生与胚胎发育有某种神奇的共性。
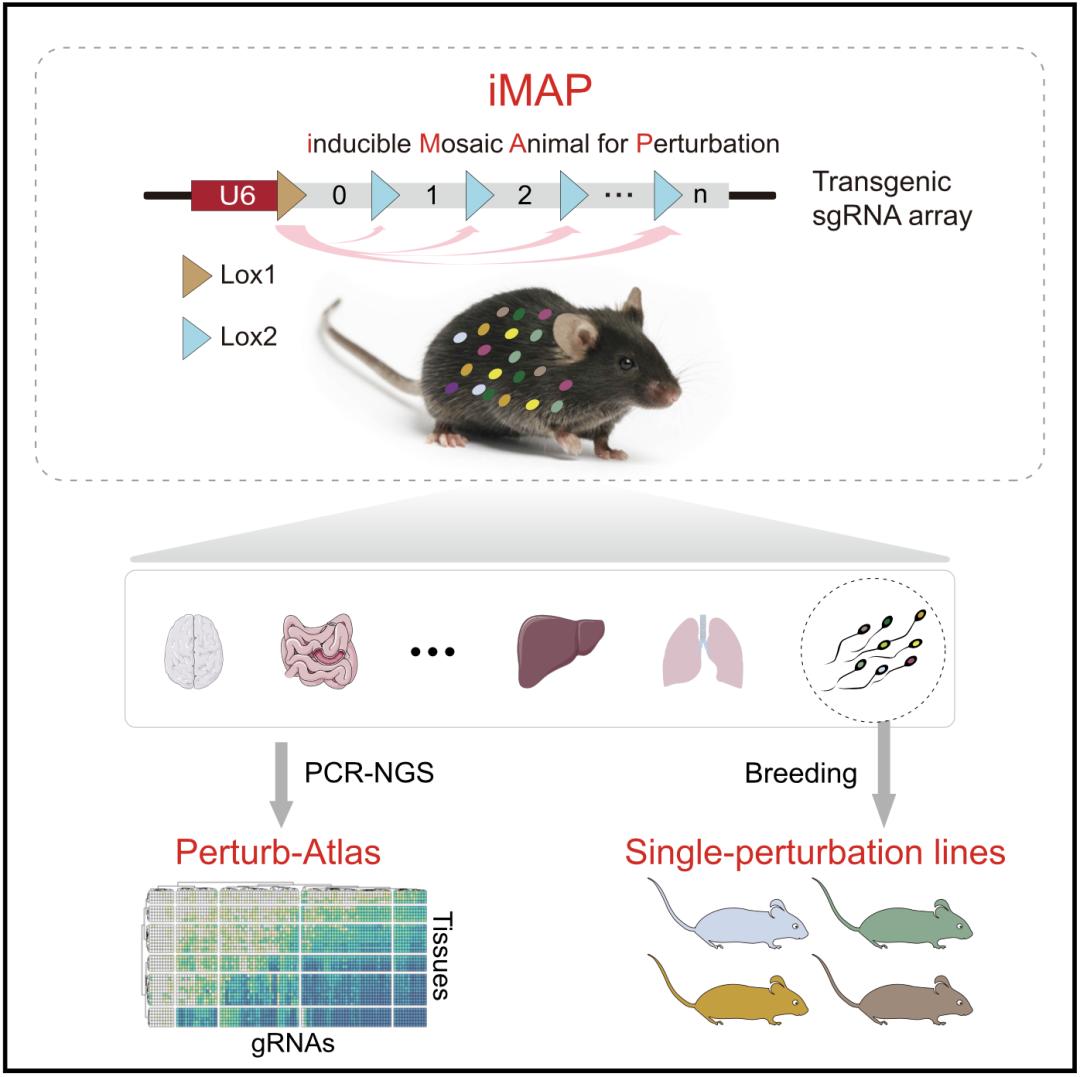
总的来说,池天课题组的的研究成果表明,iMAP作为可诱导型嵌合体鼠,可在其全身各组织中鉴定100个基因的功能,也可用于衍生100种单基因敲除品系。
另外,iMAP品系可像普通转基因鼠一样维持、分享并且反复使用。最后,iMAP与单细胞组学技术相容,从而可用于描绘单细胞扰动图谱。
如同其他新技术一样,iMAP也存在缺点,目前主要是其通量(100个sgRNA)有待提高。可以预测,iMAP将有力地推动基因解码和人类疾病的诊断治疗。
据悉,单细胞扰动图谱的领军人物Aviv Regev已与池天在酝酿发起“单细胞扰动图谱计划”。
池天课题组简介:
池天博士,上海科技大学正教授,耶鲁大学免疫生物系客座教授。聚焦基因技术的研发及其在免疫相关疾病(肿瘤、自身免疫病)的应用(Nature Comms, 2020a,b;EMBO 2020a,b;Cancer Letters 2021)。
最近发表了突破性的基因解码技术iMAP,能快速有效地进行小鼠体内CRISPR筛选(Cell 2022)。拟利用iMAP,挖掘重大疾病的关键治疗靶点,发现老药的重要新用途。
池天课题组急需认真热情具有团队协作精神的各级科研人员(研究员、博后、科研助理)加盟,熟悉肿瘤免疫/分子克隆/细胞培养/生物信息学/小鼠模型者优先。
课题组首页:https://slst.shanghaitech.edu.cn/ct/
已购买课程的朋友,
直接进入小程序收听加餐哦~
参考文献:
1.Bown, S.D.M., and Lad, H.V. (2019). The dark genome and pleiotropy: challenges for precision medicine. Mamm. Genome Off. J. Int. Mamm. Genome Soc. 30, 212–216. 1153 https://doi.org/10.1007/s00335-019-09813-4.
2.Rood, J.E., and Regev, A. (2021). The legacy of the Human Genome Project. Science 373, 1442–1443. https://doi.org/10.1126/science.abl5403.
3.Liu et al., Large-scale multiplexed mosaic CRISPR perturbation in the whole organism, Cell (2022), https:// doi.org/10.1016/j.cell.2022.06.039
4.Doench, J.G. (2018). Am I ready for CRISPR? A user’s guide to genetic screens. Nat. Rev. Genet. 19, 67–80. https://doi.org/10.1038/nrg.2017.97.
5.Kuhn, M., Santinha, A.J., and Platt, R.J. (2021). Moving from in vitro to in vivo CRISPR screens. Gene Genome Ed. 2, 100008. https://doi.org/10.1016/j.ggedit.2021.100008.
6.Jin, X., Simmons, S.K., Guo, A., Shetty, A.S., Ko, M., Nguyen, L., Jokhi, V., Robinson, E., Oyler, P., Curry, N., et al. (2020). In vivo Perturb-Seq reveals neuronal and glial abnormalities associated with autism risk genes. Science 370. https://doi.org/10.1126/science.aaz6063.
7.Rogers, Z.N., McFarland, C.D., Winters, I.P., Seoane, J.A., Brady, J.J., Yoon, S., Curtis, C., Petrov, D.A., and Winslow, M.M. (2018). Mapping the in vivo fitness landscape of lung adenocarcinoma tumor suppression in mice. Nat. Genet. 50, 483–486. https://doi.org/10.1038/s41588-018-0083-2.
8.Wang, G., Chow, R.D., Ye, L., Guzman, C.D., Dai, X., Dong, M.B., Zhang, F., Sharp, P.A., Platt, R.J., and Chen, S. (2018). Mapping a functional cancer genome atlas of tumor suppressors in mouse liver using AAV-CRISPR–mediated direct in vivo screening. Sci. Adv. 4, eaao5508. https://doi.org/10.1126/sciadv.aao5508.
9.Wertz, M.H., Mitchem, M.R., Pineda, S.S., Hachigian, L.J., Lee, H., Lau, V., Powers, A., Kulicke, R., Madan, G.K., Colic, M., et al. (2020). Genome-wide In Vivo CNS Screening Identifies Genes that Modify CNS Neuronal Survival and mHTT Toxicity. Neuron 106, 76-89.e8. https://doi.org/10.1016/j.neuron.2020.01.004.
10.Chen, Z., Arai, E., Khan, O., Zhang, Z., Ngiow, S.F., He, Y., Huang, H., Manne, S., Cao, Z., Baxter, A.E., et al. (2021). In vivo CD8+ T cell CRISPR screening reveals control by Fli1 in infection and cancer. Cell 184, 1262-1280.e22. https://doi.org/10.1016/j.cell.2021.02.019.
11.Dong, M.B., Wang, G., Chow, R.D., Ye, L., Zhu, L., Dai, X., Park, J.J., Kim, H.R., Errami, Y., Guzman, C.D., et al. (2019). Systematic Immunotherapy Target Discovery Using Genome-Scale In Vivo CRISPR Screens in CD8 T Cells. Cell 178, 1189-1204.e23. https://doi.org/10.1016/j.cell.2019.07.044.
12.Huang, H., Zhou, P., Wei, J., Long, L., Shi, H., Dhungana, Y., Chapman, N.M., Fu, G., Saravia, J., Raynor, J.L., et al. (2021). In vivo CRISPR screening reveals nutrient signaling processes underpinning CD8+ T cell fate decisions. Cell 184, 1245-1261.e21. https://doi.org/10.1016/j.cell.2021.02.021.
13.LaFleur, M.W., Nguyen, T.H., Coxe, M.A., Yates, K.B., Trombley, J.D., Weiss, S.A., Brown, F.D., Gillis, J.E., Coxe, D.J., Doench, J.G., et al. (2019). A CRISPR-Cas9 delivery system for in vivo screening of genes in the immune system. Nat. Commun. 10, 1–10. https://doi.org/10.1038/s41467-019-09656-2.
14.Wei, J., Long, L., Zheng, W., Dhungana, Y., Lim, S.A., Guy, C., Wang, Y., Wang, Y.-D., Qian, C., Xu, B., et al. (2019). Targeting REGNASE-1 programs long-lived effector T cells for cancer therapy. Nature 576, 471–476. https://doi.org/10.1038/s41586-019-1821-z.


责任编辑丨BioTalker
原标题:《《细胞》:上海科技大学池天课题组发明新型基因打靶技术,绘制世界首张“扰动图谱”丨科学大发现》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。





- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2025 上海东方报业有限公司




