- +1
猴痘病毒基因测序最新进展:指向同一源头,病毒突变数超预期
引发本轮全球猴痘疫情的猴痘病毒基因测序工作又有新进展。在此前第一份猴痘病毒基因序列草案的基础上,葡萄牙国家卫生研究所的约翰·保罗·戈麦斯(João Paulo Gomes)博士及其同事当地时间5月23日在(https://virological.org)网站上,继续公布了另外9份葡萄牙病患所携带的猴痘病毒基因组序列。
至此,全球科学家已得到了13份最新猴痘病毒基因测序结果,样本分别来自葡萄牙、德国、比利时、美国。科学家们将这些结果汇总后发现,本轮猴痘疫情暴发极有可能来自一个源头,病毒株虽然与3年前在英国发现的毒株关系密切,但与其相比,出现了平均50个单核苷酸多样性位点(SNPs)突变。考虑到正痘病毒作为DNA病毒的变异率,这样的变异程度超出了科学家的预期。
5月以来,罕见而不寻常的猴痘疫情在全球多个国家暴发。世卫组织5月24日通报,目前已有19国已报告131例猴痘确诊和106例疑似病例,而在本轮疫情暴发前,猴痘病例主要在非洲中部和西部地区热带雨林散发。5月23日,世卫组织主要顾问大卫·海曼(David Heymann)表示,有分析认为欧洲此前两场狂欢派对中大规模性行为或是本轮猴痘蔓延的主要原因。
约翰.保罗.戈麦斯介绍,最新公布的9份基因组序列检测结果来自2022年5月15日和17日采集的9例葡萄牙患者的标本,团队对患者的皮肤渗出液进行病毒DNA采样,而后在Illumina平台上,通过高通量鸟枪法宏基因组测序法对病毒基因组进行了(2 × 150 bp )的双端测序,测序覆盖深度从38倍到508倍不等(平均为201倍),相较于第一份样本使用的Nanopore平台、平均覆盖深度约7倍,测量的精确度有了较大提高。
团队将新测序的基因组整合到猴痘病毒基因进化树中,并与美国公布的病毒基因组序列(MPXV_USA_2022_MA001)相比较,得出观察结论称,这次多国猴痘暴发很可能只有一个源头,迄今为止公布的所有病毒基因序列都紧紧聚集在一个分支上。
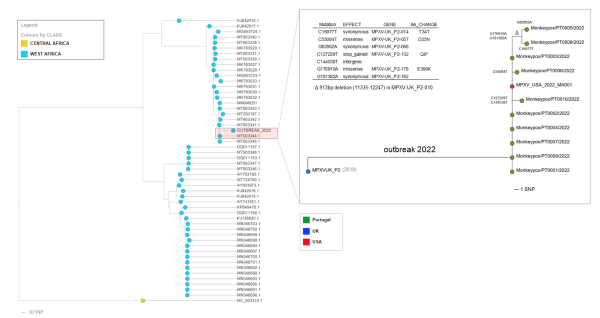
图一:葡萄牙约翰.保罗.戈麦斯团队绘制的猴痘病毒基因进化树
团队表示,通过对样本基因组序列在病毒基因进化树上的定位,可以确认,本轮暴发的猴痘病毒属于较温和的西非分支,与2018年和2019年从尼日利亚向多个国家(英国、以色列和新加坡)输出的猴痘病毒关系最为密切。
然而,值得担忧的是,此次暴发的猴痘病毒基因组与2018-2019年病毒基因组相比,平均差异了50个单核苷酸多态性位点 (SNPs,主要是指在基因组水平上由单个核苷酸的变异所引起的 DNA 序列多态性。它是生物可遗传的变异中最常见的一种。)考虑到正痘病毒的预计替换率,这远远超出了人们的预期。
在此之前, 英国爱丁堡大学的演化生物学家安德鲁·兰博特(AndrewRambaut)表示,“我们曾估计天花病毒(猴痘的近缘病毒,同属正痘病毒属)的基因组每年每个位点大约有1x10-5个替换,从而造成病毒平均每年1-2个核苷酸变化。这是估计的高端。”
此外,该团队在暴发集群中发现了病毒基因微进化的第一个迹象,即出现了7个单核苷酸变异位点(SNPs),其导致3个后代分支(图1),其中一个分支涉及2个基因序列(PT0005和PT0008)的亚集群(由2个SNPs支持)。值得注意的是,这2个序列还出现了基因移码缺失的情况。
“基因丢失事件在猴痘流行的中非环境中已经被观察到,并被假设与人与人之间的传播有关。”约翰.保罗.戈麦斯说。他同时表示, 这种微进化的情况也表明,基因组测序可能提供足够的分辨率来跟踪当前疫情背景下的病毒传播。
宿主APOBEC3编辑机制或引发病毒变异
对于最新猴痘病毒出现这种单核苷酸变异的原因, 目前多数科学家认为,这是由宿主体内的APOBEC3蛋白质编辑机制引发。
戈麦斯分析,“我们不能放弃这样的假设,即猴痘病毒基因进化树上的分叉支,是由病毒宿主体内的APOBEC3编辑机制引起的病毒进化跳跃(导致病毒高度突变) 。”
APOBEC3蛋白酶在人和一些哺乳动物体内存在,能起到防御病毒的作用。其主要的抗病毒机制是它能够在病毒单链DNA的复制过程中,使胞嘧啶碱基脱去氨基,变成尿嘧啶碱基,当另一条链合成时,尿嘧啶碱基与腺嘌呤碱基配对,从而产生对病毒致死的G→A高突变现象。
此前,在美国一研究团队公布5月18日在马塞诸塞州确诊的猴痘病患携带病毒的基因测序数据后,安德鲁·兰博特就将美国样本(MA001)、葡萄牙第一份样本(PT0001)与三年前的英国样本(UK_P3)比较,即观察到,从2018年到2022年,病毒基因组出现单核苷酸多态性位点的差异具有极大的倾向性, 除了2个例外情况,所有的G → A 变异都表现为 GA → AA, 所有的C → T 变异都表现为TC → TT,这似乎都是APOBEC3编辑模式偏好的单核苷酸变异。
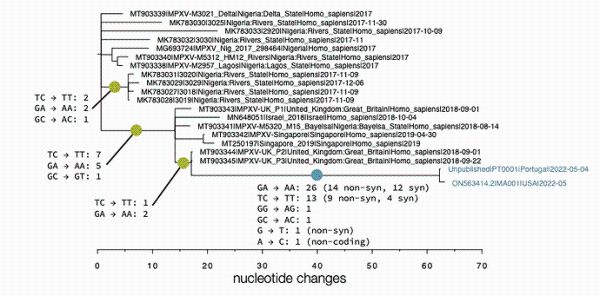
图二:英国爱丁堡大学的演化生物学家安德鲁·兰博特标记的猴痘病毒基因进化树
兰博特认为,在大多数情况下,病毒这种随机的高度突变会造成蛋白质功能的改变和破坏,使病毒失去活性。然而,有时APOBEC3编辑机制并不能对病毒产生足够的攻击从而使其失活,病毒会继续复制和传播。虽然导致2022年猴痘爆发的病毒株在进化树上分支异常长,但根据这个模型可以预测,所有或大部分突变即有可能发生在一轮复制中。
在葡萄牙戈麦斯团队新的测序结果公布后, 兰博特评论说,“所有新出现的单核苷酸多态性位点在二核苷酸环境中也都是TC→TT或GA→AA。这都支持了大多数单核苷酸突变是宿主蛋白酶编辑的结果,而不是DNA聚合酶复制错误的产物。
不过, 戈麦斯对APOBEC3编辑机制也提出了一些疑问,他认为,以往的研究结果显示,在APOBEC3编辑机制下,都是在基因组局部区域中,出现小片段的C → T和G→A倾向性变异,而不是像这里观察到的那样,扩散到一个广泛的基因组区域——长度达到了150kb。此外,考虑到APOBEC3 的作用机制,它需要在DNA处于单链状态下才能对其产生作用。
“这令人费解。”他说。
对此, 兰博特猜测,这可能是病毒基因组在进行多次复制时,出现了将DNA的两条链都暴露出来的情况,从而使得APOBEC3蛋白酶能够对两条链上的碱基产生作用。
美国圣地亚哥J.Craig Venter Institute的生物信息学博士 Anna Maria Niewiadomska则对不同宿主的APOBEC3编辑机制作出进一步阐释。她称, APOBEC3编辑非常依赖于宿主物种。与啮齿动物相比,人类拥有更多的APOBEC3蛋白酶库(A3A、A3B、A3C、A3D、A3F、A3G和A3H),而啮齿动物(大多数)只有一个双结构域A3蛋白酶。不同的蛋白酶倾向于在不同的环境中突变C,并且可能有不同的细胞定位。例如,人类A3G会优先突变CCC > CCT中的C,而小鼠A3优先发生突变TYC > TYT中的C。
兰博特对此回应说,“我不认为目前的病毒基因组数据能让我们区分宿主,但葡萄牙最新的基因组序列显示病毒最新变异确实发生在人类身上,而基因进化树上的黄色点(图二)意味着变异也曾发生在非人类动物身上。”





- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




