- +1
中科院微生物所团队利用深度学习方法,合成216种新型抗菌肽,超八成有抗菌活性
原创 果壳硬科技 我是科学家iScientist
2022年3月3日,中国科学院微生物研究所在Nature Biotechnology发表论文,利用深度学习方法,构建了从人类肠道微生物组挖掘潜在新型抗菌肽的模型,总计挖掘并合成了216种潜在的新型抗菌肽,其中83.8%具有抗菌活性,对多重耐药革兰氏阴性菌具有较强的抑菌能力,同时在动物感染模型中验证部分抗菌肽具有非常好的体内活性和安全性,可能成为应对如今抗微生物药物耐药性危机的一大潜力策略。
耐药性危机与
新型抗菌剂“抗菌肽”开发的挑战
世界卫生组织近年的一份报告[1]指出,抗微生物药物耐药性已经成为人类健康的直接威胁。在资源有限的地区,在新生儿和幼儿等弱势群体中,抗微生物药物耐药性的影响最为严重,细菌性肺炎和血流感染是5岁以下儿童死亡的主要原因,大约30%患败血症的新生儿死于对多种一线抗生素耐药的细菌感染[2]。
然而近几十年来,几乎所有投入市场的新型抗生素都是1980年以前发现的抗生素药物类别的变体。因此,世界迫切需要开发新的抗菌疗法。报告[1]中列出了正在开发的27种非传统抗菌素,其中就包括了能抵御多重耐药菌同时不易产生耐药性的“抗菌肽”(AMPs),它被认为是极有发展前景的一类新型抗菌剂。
相对于极易诱导细菌产生耐药突变的传统抗生素,抗菌肽直接作用于细菌细胞膜来杀灭细菌,这意味着细菌必须改变相当部分的基因直至改变膜的结构才能防御抗菌肽的进攻,而这几乎是不可能的。同时,也因为真核细胞膜与细菌细胞膜电性的不同,使得抗菌肽在杀灭细菌的同时,能够很大程度上保证动物体和人体的安全性。加之它经过简单的后期催化修饰或者不经任何修饰就可以直接发挥作用,因此,抗菌肽具有非常大的开发和应用潜力。
我们知道,不少耐药病原菌本身就是肠道共生微生物。这种紧密的共生关系暗示人体肠道微生物可以产生抑制这些耐药病原菌的化合物,维持人体的微生态平衡;人体微生物中天然存在着错综复杂的互作网络,其中必然也蕴含着大量能够编码产生抗菌肽分子的功能序列[3]。然而,到目前为止,由于其序列短、多样性高、相似性低的特点,抗菌肽的发现在很大程度上仍然是实验驱动的,缺乏可靠的生物信息学方法来在海量的微生物组数据中高效、高通量地挖掘新型抗菌肽,限制了这类潜在药物分子的开发[4]。
值得高兴的是,中国科学院微生物研究所3月3日发布的利用深度学习识别人类肠道微生物组的抗菌肽的文章,采用自然语言学习(NLP)的多种神经网络方法,实现了抗菌肽挖掘模型的构建和优化;通过该预测模型在大规模微生物组(1万余样本)中的应用,总计挖掘并合成了216种潜在的新型抗菌肽,并通过实验验证了其抗菌性能与安全性。
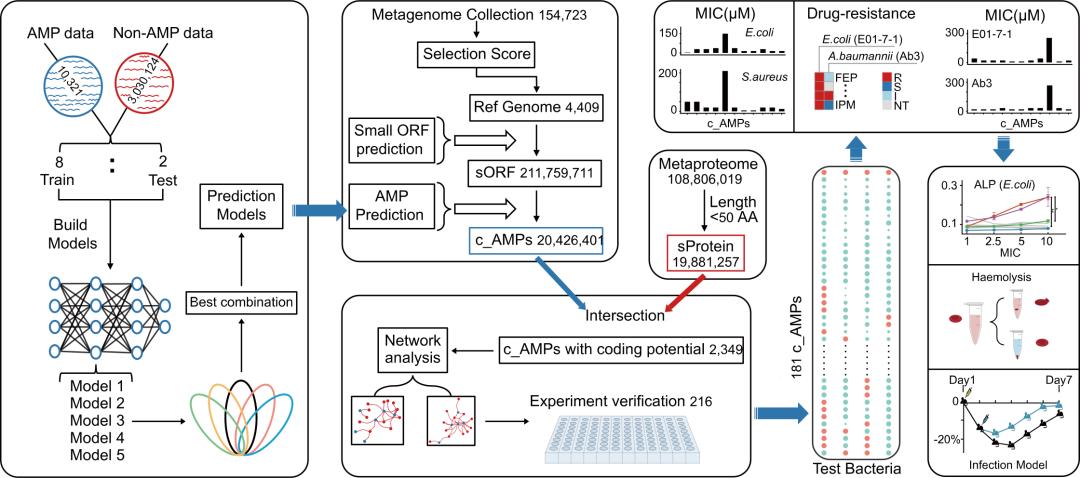
研究流程示意图。收集抗菌肽序列用于构建优化预测模型(左图);微生物组中挖掘潜在的抗菌肽序列,通过网络分析进一步过滤假阳性序列,将最终得到的新型抗菌肽进行化学合成和初步实验验证(中间部分);将效果最优的肽进行后续研究,包括针对多重耐药菌的药效测试在内的多项研究(右图) | 参考文献[5]
抗菌肽预测模型构建
及序列鉴定和筛选
在这一研究中,作者选取了三种自然语言处理神经网络模型用于预测鉴定抗菌肽。在测试集中,预测模型的精确度(Precision)达到了91.31%,超过了其他已知的抗菌肽预测工具。
他们应用微生物组数据预测出长度在50个氨基酸以下的序列,通过结合表达数据、菌丰度关联分析、假阳性序列排除后后最终得到241条潜在抗菌肽序列,其中216条被成功合成。
“我们基于深度学习的预测模型不同于根据序列相似性、氨基酸组成或性质建立的模型,这一模型不仅能够识别出在以往研究中尚未被挖掘的新型AMPs,还让我们更有可能获得具有不同结构、不同作用机制的AMPs。” 论文共同第一作者张雨薇向果壳介绍道。
实验结果证明了作者对预测模型的推论:对所有成功合成的序列进行初步活性筛选,其中181条显示出抗菌活性(阳性率大于83%),且绝大多数与已知的AMPs的序列同源性(identity)低于40%,作用机制也不局限于某一类特定的机制。
“新型AMPs能够拓展我们对微生物组数据中编码活性代谢产物序列的了解,也能在一定程度上对目前严峻的细菌耐药性问题提供解决思路。”张雨薇博士表示。
潜在抗菌肽的抑菌性能评估
研究者挑选出181条潜在抗菌肽序列中对抗革兰氏阴性菌最有效的11条,进行了新一轮菌的活性测试实验,包括粪肠球菌、金黄色葡萄球菌、肺炎克雷伯菌、鲍曼不动杆菌、绿脓杆菌、阴沟肠杆菌和大肠杆菌。
实验结果发现,这11条肽中对单个菌以及对临床分离得到的多重耐药菌大都具有强劲的抑制能力,大部分MIC值远小于50µM。
通过溶血实验和细胞毒性实验,从11条潜在抗菌肽中筛选出3条溶血和细胞毒性相对较低的小肽进行后续的体内实验测定。首先对这3种肽进行了时间杀伤性试验,测定了各自的最佳作用时间。接着在肺部感染肺炎克雷伯菌的小鼠模型中进行了体内实验,测定了小鼠体重恢复曲线以及给药后肺部组织中肺炎克雷伯菌遗留量,相对于非治疗组,治疗组的小鼠肺部组织菌遗留量和体重恢复速度的表现都明显更为优秀。
潜在抗菌肽的作用机制探究
作者通过碱性磷酸酶(ALP)、1-N-苯基萘胺 (NPN)、3,3′-二丙基噻二碳菁碘 [DiSC3(5)]和碘化丙啶(PI)实验在11条潜在抗菌肽中确定了部分肽的作用机制分别为细胞膜和细胞壁。同时仍有部分肽没有观察到在以上两个部位有明显作用效果。
接着选取了作用于细胞膜上且抑菌效果最明显的肽应用透射电子显微镜进行直接观察,可以看到与肽共培养的大肠杆菌细胞膜有明显的破裂现象。
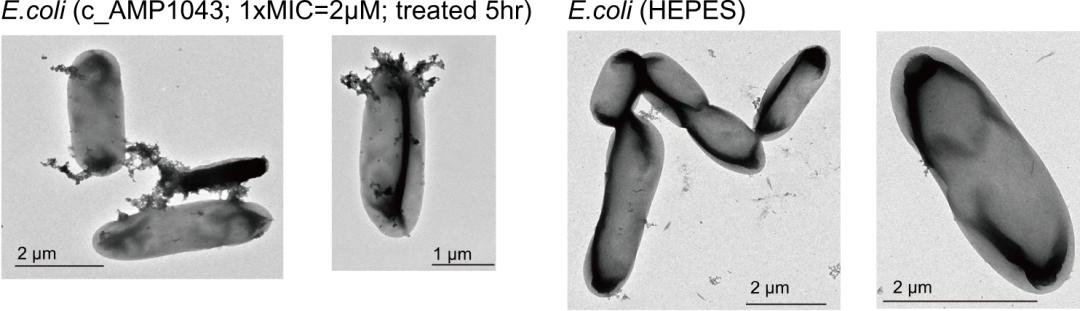
透射电镜观察结果 | 参考文献[5]
值得注意的是,作者还对同一个肽继续进行了对大肠杆菌的抗性诱导研究,经过长达30天的连续传代培养,并未检测到任何耐药性。
最后,对11条肽和在181条肽中另随机选择的肽进行了圆二色谱的测定,结果表明在11条肽处于脂质环境下时,超过一半的肽由alpha-螺旋结构主导;而在随机挑选的肽中这一现象并不明显。alpha-螺旋结构主导的肽可能有更强的抑菌活性。
能做到什么?想做到什么?
该研究以人体肠道微生态为切入点,通过结合微生物组大数据和最新的深度学习模型对人体肠道微生物中蕴含的活性代谢产物进行了高效挖掘,成功获得了在体内、体外实验中均表现出良好抗耐药菌活性的抗菌肽。
然而,预测模型的建立和预测能力与可收集的数据量密切相关,作者的模型在预测抗菌肽的抗菌活性方面具有良好表现,而对抗菌肽其他方面数据的预测在一定程度上受已有数据量和数据质量的限制。
“例如,大多数抗菌肽仅记录了对大肠杆菌和金黄色葡萄球菌的抗菌活性,因此,目前还难以针对抗菌肽作用的菌种种类或其他指标建立准确的预测模型。相信随着数据量和检测指标的增加,我们将能够建立更全面的预测模型。”张雨薇博士生介绍道。
后续研究中,研究团队表示一方面将对已获得的抗菌肽进行优化,逐步提高其成药性;另一方面也会尝试将抗菌肽的活性范围优化到针对其他病原菌上。
更重要的是,随着人们对微生态研究的不断深入和测序数据的不断积累,这一研究策略将能够应用于不同环境下对多种活性分子的挖掘,也即研究者将能够对更多部位的人体微生物进行挖掘,并将关注的功能从抗菌活性延伸到代谢疾病、免疫性疾病的治疗等领域。
目前,团队正在针对已有的活性好、安全性高的抗菌肽进行优化,希望能够进一步提高药代性能。
“我们也欢迎感兴趣的生物医药公司和我们一起合作,开发新的抗菌药物应用于耐药菌治疗。另外,利用相同的流程我们还发现了具有调节免疫或者改善代谢功能的多肽,也在做相应的机理研究和动物实验。除此之外,还有海量的基因组数据可以做针对不同目的的进一步挖掘,后续的这些工作都具有非常大的转化价值,我们希望能与生物医药产业开展更多合作研发和转化。”研究团队表示。
致谢
感谢陈义华研究员的支持和张雨薇博士生对本文的建议。
参考文献
[1] World Health Organization. 2020 antibacterial agents in clinical and preclinical development: an overview and analysis[J]. 2020 antibacterial agents in clinical and preclinical development: an overview and analysis, 2021.
[2] Folgori L, Ellis S J, Bielicki J A, et al. Tackling antimicrobial resistance in neonatal sepsis[J]. The Lancet Global Health, 2017, 5(11): e1066-e1068.
[3] Walsh, C. T. Insights into the chemical logic and enzymatic machinery of NRPS assembly lines. Nat. Prod. Rep. 33, 127–135 (2016).
[4] Spänig, S. & Heider, D. Encodings and models for antimicrobial peptide classification for multi-resistant pathogens. BioData Min. 12, 7 (2019).
[5] Ma, Y., Guo, Z., Xia, B. et al. Identification of antimicrobial peptides from the human gut microbiome using deep learning. Nat Biotechnol (2022). https://doi.org/10.1038/s41587-022-01226-0
作者:竹子
编辑:酥鱼
排版:尹宁流
题图来源:图虫创意
研究团队
中国科学院微生物研究所王军课题组马越、夏彬彬,陈义华课题组郭正彦、张雨薇为本文的共同第一作者,王军研究员和陈义华研究员为共同通讯作者。
本研究受到了中科院战略先导项目“病原体宿主适应与免疫干预” 和战略生物资源计划、科技部重点研发、国家自然科学基金委杰出青年基金项目(陈义华)、面上项目和“糖脂代谢的时空网络调控”重大研究计划培育项目,以及北京市科技新星项目的支持。
论文信息
发布期刊 《自然-生物技术》Nature Biotechnology
发布时间 2022年3月3日
论文标题 Identification of antimicrobial peptides from the human gut microbiome using deep learning
(DOI:https://doi.org/10.1038/s41587-022-01226-0)
原标题:《中科院微生物所团队利用深度学习方法,合成216种新型抗菌肽,超八成有抗菌活性》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。




- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




