- +1
【重磅综述】 “路漫漫其修远兮,吾将上下而求索”,DNA 6mA修饰研究全纪录
原核生物中一种广泛存在的DNA修饰——6mA,近年来被发现也存在于高等真核生物基因组当中。尽管6mA在原核生物中已经研究的很充分,但是6mA在真核生物中的功能和调控机理的探索才刚刚起步。
2021年8月17日,贝勒医学院吴涛(Tao P. Wu)团队在Protein & Cell发表综述文章The Exploration of N6-Deoxyadenosine Methylation in Mammalian Genomes ,详细总结了近年来关于真核生物,特别是哺乳动物中DNA 6mA的检测方法,以及调控和功能研究的进展。

DNA甲基化在基因表达调控,染色质结构控制,人类疾病,衰老和肿瘤发生等很多生物学过程中都扮演着重要角色【1-3】。尽管DNA 4mC,5mC和6mA很早就被发现存在于原核生物和单细胞真核生物基因组,但根长一段时间内5mC及其衍生物(5hmC,5fC,5caC)被认为是哺乳动物中仅有的DNA表观修饰【4】。经过几十年的探索,特别是2015年以来的一系列研究确定了6mA也存在于包括哺乳动物在内的多种高等真核生物基因组当中【5-8】。
原核生物中的6mA
DNA 6mA在上世纪50年代第一次在Bacterium coli中被发现【9,10】。进一步的研究发现6mA是细菌基因组中一种普遍的修饰【11】。6mA随后被证实在限制修饰系统(R-M system)中发挥重要作用【12-14】。这些研究也表明在大肠杆菌(E. coli)中只有小部分的6mA是用于限制修饰系统的。余下大部分6mA的功能直到Dam甲基化转移酶发现后才慢慢浮出水面【15】。Marinus等发现Dam突变的细菌对UV照射和mitomycin C处理更敏感,Dam突变株的DNA含有更多的单链DNA断裂,显示6mA能防止DNA断裂。之后的研究表明6mA在DNA复制起始【16】,DNA错配修复【17】和基因表达调控【18,19】中也有重要作用。
真核生物6mA的发现
除了原核生物,DNA 6mA也很早被发现在单细胞真核生物中存在【20,21】。但是直到近年来才被发现也存在于后生动物和高等植物当中。利用高效液相色谱-质谱联用技术和单分子实时测序,6mA在2015年被发现存在于绿藻【5】,线虫【7】和果蝇【6】基因组,并在2016年首次被发现存在于小鼠干细胞基因组【8】,之后也相继被发现存在于猪【22】和人【23】等哺乳动物的基因组中。
真核生物6mA的检测方法
除了液相色谱-质谱联用技术(LC-MS/MS)和单分子实时测序技术(SMRT-seq),基于抗体的免疫印迹 (Immunoblotting),免疫荧光(IF)和免疫沉淀共测序(DIP-seq)等方法也被用于6mA的基因组定位和定量研究。但是6mA在大多数报道的哺乳动物基因组中的丰度很低,导致哺乳动物 6mA 的准确检测极具挑战性。因此,文章总结了目前用于检测真核生物中6mA的方法和可能被应用于进一步研究6mA的基因组分布的新方法。
基于质谱的检测方法
目前,液相色谱-质谱联用技术的灵敏度能达到千万分之一,还能区分开DNA 6mA和RNA m6A或者其他DNA修饰,是定量极低丰度6mA的金标准【24,25】。但是由于6mA在细菌,支原体等微生物中丰度很高,基于质谱的检测方法必须排除微生物(支原体、细菌等)污染的影响,避免产生假阳性结果。因此,准备样品时确保无污染对6mA的研究是必须的。另外,最近的研究表明质谱实验中用于水解DNA的酶(通常从细菌中纯化而来)也可能带有细菌的6mA而产生假阳性结果【26】;通过添加空白对照的方法可以很容易排除这种假阳性【26,27】。
抗体依赖的检测方法
相对于液相色谱-质谱联用技术,抗体依赖的方法如免疫印迹和免疫荧光的灵敏度低但是简单易用,所以被广泛的应用于研究6mA。但是由于现有的抗体无法区分DNA 6mA和RNA m6A以及 m6Am,而且通常RNA m6A的丰度远高于DNA 6mA,因此,去除DNA中的RNA污染对抗体依赖的检测方法是必要的。抗体还可用于DIP-seq来检测6mA在基因组中的分布。但是抗体的特异性需要对每种实验系统单独优化,6mA丰度对于免疫沉淀的富集效果很重要,6mA修饰的寡核苷酸spike-in实验表明6mA的丰度需要到10-15ppm以上,(多克隆抗体)免疫沉淀才能达到富集6mA十倍以上的效果【8】。因此,6mA DIP-seq在DNA丰度太低情况下会产生很多的非特异性信号【28】。最近的研究表明6mA DIP-seq在类滋养层干细胞中用Input,IGG或者全基因组扩增(WGA)产物作为对照得到类似的结果,说明6mA DIP-seq在6mA丰度高于一定的阈值时, 信号是足够特异的【27】。
非抗体依赖的检测方法
为了消除上述抗体的非特异性信号和验证基于抗体的实验结果,不依赖抗体的检测方法对于6mA的研究非常重要。第一种不依赖抗体的方法是利用甲基化敏感的限制性内切酶切割DNA来检测6mA【5,29,30】。但是甲基化敏感的限制性内切酶依赖于特定的DNA基序(GATC,CATG等)。但是现有的6mA DIP-seq和SMRT-seq没有在哺乳动物基因组中检测到类似的6mA基序【8,23,31,32】,因此这类方法很难用于检测哺乳动物的6mA。
另一种不依赖抗体的检测方法是第三代测序技术,SMRT-seq通过DNA 聚合酶复制时动力学特性的变化来检测6mA【33】。SMRT-seq已经被成功的应用于哺乳动物和植物的6mA检测【8,23,32-35】。但是也有报道显示SMRT-seq在6mA丰度很低的时候会产生假阳性【28,32】。因此,极低丰度的6mA的SMRT-seq检测需要富集6mA和很高的测序覆盖度【32】。另外一种极具潜力的三代测序技术——纳米孔测序也被报道能用于检测6mA,但是目前还只被用于检测原核生物或者单细胞真核生物的6mA【36-39】。
此外,很多实验室也在积极开发6mA检测的化学生物学方法。Hong等发现Ag+会导致引物在6mA位点延伸终止,但不会导致未甲基化的腺嘌呤位点延伸终止,这可以用来选择性地检测6mA【40】。然而,由于无法指数化扩增DNA片段,这种方法难以用于6mA的全基因组检测。另外,Nappi等报告了一种通过光结合后的生物素连接选择性地标记6mA的方法。这种标记的6mA可以接着被富集而进一步通过qPCR或测序进行定量【41】。Nappi等进一步证明,这种方法的富集效果与基于6mA抗体的免疫沉淀法相当。另外,Mahdavi-Amiri等开发了亚硝酸盐测序。亚硝酸盐测序法利用亚硝酸钠和乙酸对未甲基化的腺嘌呤进行重氮化和脱氨,而甲基化的腺嘌呤不能完成脱氨【42】。因此,未甲基化的腺嘌呤在PCR后将转化为鸟嘌呤,而甲基化的腺嘌呤将保持为腺嘌呤。通过计算每个碱基的相对A->G突变率,可以在单碱基分辨率下区分和量化6mA。然而,这种方法目前需要高比例的6mA。
6mA的调控
哺乳动物中 6mA 可能的甲基转移酶
由于 6mA 的低丰度和模式功能系统的缺乏,哺乳动物中6mA的甲基转移酶仍然是目前最具挑战性的核心问题。尽管如此,一些团队已经报道了几个可能的6mA甲基转移酶,比如N6AMT1【23】,METTL4【43】。但是研究表明N6AMT1的甲基转移酶活性胶质母细胞瘤中在无法重复【31】。此外,结构生物学和生物化学研究显示N6AMT1是蛋白质eRF1的甲基转移酶而无法结合和催化DNA甲基化【44】。METTL4是线虫和四膜虫中鉴定的甲基转移酶的同源物。Kweon等通过基因过表达和敲除实验首次报道了METTL4是人源细胞系和小鼠胚胎干细胞中核DNA的甲基转移酶【43】。METTL4后来也被鉴定为线粒体DNA的6mA甲基转移酶【45】。另外,有趣的是, MD Anderson癌症中心Xiaodong Cheng实验室Woodcock等报道了METTL3-14复合物——众所周知的RNA m6A的甲基转移酶复合体也可以在体外甲基化DNA【46】,但是目前还不清楚METTL3-14复合物是否也能在细胞内甲基化DNA。总的来说,METTL4目前还缺乏METTL4催化核基因组DNA甲基化的体外证据。芝加哥大学Chuan He实验室已经体内体外同时验证了METTL4对线粒体DNA的活性。与此同时,搜索其他可能的甲基转移酶和确定不同底物对甲基转移酶的影响也很重要。
哺乳动物生理性6mA的其它可能来源
哺乳动物6mA丰度低且缺乏明确的甲基转移酶,这促使科学家开始考虑除甲基化酶之外的可能性,比如6mA 可能是在 DNA 复制和 DNA 损伤修复过程中被 DNA 聚合酶错误掺入,而不是通过甲基转移酶直接甲基化DNA。2004年Charles等的研究利用6mA脱氧腺苷处理C2C12细胞首先证实了6mA可以被整合进入基因组【47】。Musheev等最近的研究显示哺乳动物细胞中极低的6mA来源可能是降解的RNA m6A单体通过代谢产生游离的N6-methyl-dATP,进而被DNA聚合酶整入基因组【48】。汪海林团队进一步实验表明DNA 6mA在细胞周期中的G1期富集,且6mA的富集依赖于DNA聚合酶lambda——一种主要参与NHEJ DNA修复过程的不依赖模版的DNA聚合酶,说明6mA可能与NHEJ DNA修复通路相关【49】。虽然这两项研究都没有在各自的系统中检测到甲基化转移酶催化形成的6mA,但是同时Musheev等的研究发现多数细胞中内源的6mA单体N6-methyl-dATP的丰度很低甚至无法被质谱检测到,汪海林团队的研究也显示N6-methyl-dATP并不是高保真DNA聚合酶偏好的底物。此外,目前还不清楚错误掺入机制能否解释此前的研究发现的6mA在一些环境刺激下高度动态的特性。6mA甲基化转移酶也有可能只在特定的条件或者环境中才有活性,因此,无论是证明还是排除6mA甲基转移酶的直接甲基化反应,都需要更多的探索。
6mA的识别
鉴定能识别6mA的蛋白(阅读器)对全面了解6mA在真核生物中的表观遗传学功能非常重要。原核生物中,MutH和SeqA能读取半甲基化的GATC序列来促进错配修复和防止过早复制【16,17】。中科院陈大华团队此前报道了Fox家族蛋白Jumu能识别6mA来调控胚胎基因表达,显示了6mA在果蝇胚胎发育中具有重要功能【50】。在线粒体中,6mA被报道能招募SSBP1,但是下游的功能仍不清楚【51】。何川团队报道了6mA能阻止TFAM结合线粒体DNA,进而抑制线粒体DNA的转录【45】。然而,哺乳动物细胞中核DNA 6mA的读取器仍然不清楚。耶鲁Andrew Xiao实验室和清华李海涛团队最近的研究阐述了6mA 通过抑制SATB1在SIDD区的结合,从而调控染色质状态并可能影响胚胎早期发育进程的机制【27】。这项研究显示不同于通常的表观遗传修饰,6mA可能主要通过抑制其他蛋白的结合DNA来实现其功能。当然也可能有其他未发现的蛋白能作为6mA的阅读器来帮助6mA实现功能。
6mA的去甲基化酶
不像哺乳动物6mA甲基转移酶的鉴定,自6mA在 小鼠胚胎干细胞中的发现以来,对 6mA 去甲基化酶的研究已经取得了许多进展。几项利用遗传和生化方法的研究表明,ALKBH1 可以作为哺乳动物基因组中的 DNA 6mA 去甲基化酶发挥作用【8,23,31,52,53】。ALKBH1很可能是通过将甲基羟基化为羟甲基而催化6mA去甲基化。这个不稳定的羟甲基可以以甲醛的形式自发释放而实现直接去甲基化,这与多步骤的5mC去甲基化过程不同。这些酶学和结构实验显示,ALKBH1更倾向于结合“泡泡状”隆起的DNA,而不是普通·的单链或双链DNA,可能是由于缺乏自主的碱基翻转活性【52,53】。虽然ALKBH1去甲基化的机制已经有了初步的阐述,但是也有一些研究没有观察到ALKBH1的去甲基化酶活性【49,54】,因此还需要更多的研究来最终确定ALKBH1的6mA去甲基化活性。ALKBH4,果蝇和线虫6mA去甲基化酶的同源蛋白,也被报道可以作为哺乳动物的去甲基化酶【43】,但是目前还没有其他研究能重复此结果。因此还需要更多的研究来确定是否ALKBH4是另一个6mA的去甲基化酶。
6mA在哺乳动物中的生物学功能
尽管6mA对原核生物的复制,转录,和限制修饰系统都至关重要。哺乳动物中6mA的生理和病理功能还十分不清楚。尤其是对于极低丰度的6mA是否真的具有重要的生物学意义的还存在争论。但是,最新的一些证据表明6mA对于细胞应对一些环境刺激比如低氧条件有重要的功能【31,45】。此外,6mA也在胚胎发育,肿瘤发生中具有重要的功能【8,27,31】。
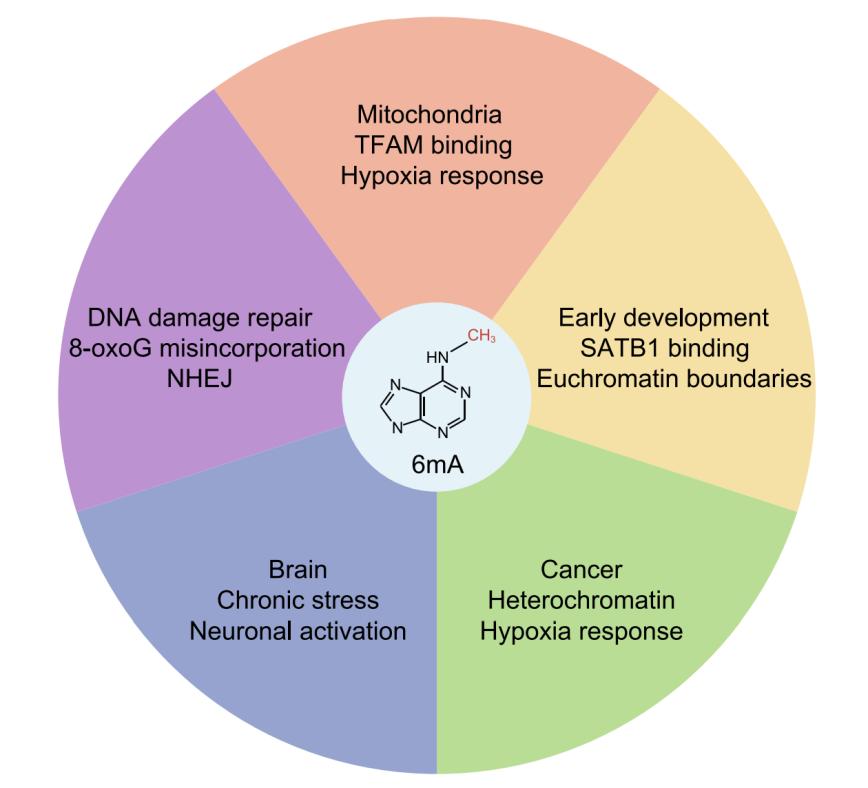
早期胚胎发育中的6mA
哺乳动物6mA是首先在小鼠干细胞中被发现,富集于H2A.X区间,并证实具有转录抑制的功能【8】。6mA在mESCs中的积累导致体外胚胎体分化过程中细胞命运决定的不平衡。同样地,研究发现6mA在斑马鱼和猪的早期胚胎发育中是被动态调控的【22】。但是关于6mA是否对胚胎发育有直接的作用还不清楚。Andrew Xiao和李海涛团队的研究首次证实了6mA在小鼠ESC-to-TSC转分化中起到关键作用【27】。研究发现在从mESCs过渡到类小鼠滋养细胞干细胞的过程中升高6mA。基因组分析显示,6mA富集在SIDD(染色质易开链)区域,并且发现6mA积累在异染色质边界,通过阻止SATB1染色质结合来限制异染色质的扩散。此外,6mA-SATB1的拮抗作用对细胞系和小鼠模型中滋养层细胞的发育至关重要。6mA是否在其他物种比如人类的胚胎发育中也具有类似的功能是一个值得关注的问题。
大脑中的6mA
除了6mA在早期发育中的功能外,研究者们还做了许多努力来研究6mA在哺乳动物大脑中的作用。Yao及其同事发现,在暴露于慢性压力的小鼠中,前额皮层(PFC)的6mA水平从6.6 ppm增加到25.5 ppm【55】。类似的,另一项研究表明6mA在体外诱导活性的初代皮层神经元中水平上升,该研究还调查了成年小鼠边缘下前额叶皮层(ILPFC)中可被恐惧消除学习选择性激活的神经元中的6mA水平【56】。然而,DpnI-seq发现了超过200万个6mA位点,这远比免疫沉淀测序和质谱方法在PFC中估计的6mA 数量要多。可能是由于不同的背景、应激类型或检测方法而观察到这样的差异。总的来说,这些研究表明,6mA在哺乳动物的大脑中可能具有重要的功能,特别是前额皮层。一个重要的问题是6mA是否在病理情况下起作用,如神经退行性疾病。
癌症中的6mA
虽然5mC在癌症发展过程中调节转录的关键作用已得到公认【57】,但其他类型的DNA修饰,如6mA,如何调节肿瘤的发生仍是未知数。谢琦和吴涛等报道,6mA在人类胶质母细胞瘤中高度上调并且靶向6mA去甲基化酶ALKBH1有望成为治疗该类癌症的新策略【31】。基因组分析显示,在胶质母细胞瘤干细胞(GSCs)中,6mA主要与异染色质组蛋白修饰H3K9me3共定位。为了研究6mA在胶质母细胞瘤中的功能,谢琦等敲除了6mA的去甲基化酶ALKBH1,并观察到6mA和H3K9me3水平的增加,以及对GSCs生长的抑制。进一步的分析表明,对ALKBH1调控的6mA在关键的致癌通路中明显富集,其中包括缺氧反馈通路。6mA随后也被报道在人类食管鳞状细胞癌中升高【58】。相反,也有研究报告称原发性胃癌、肝癌和肺癌的DNA 6mA水平相对于正常组织下降【23,59】。可能6mA的表达、调控和功能在不同类型的癌症中有所不同。需要进一步研究以阐明6mA在癌症中的功能和调节机制。
线粒体中的6mA
线粒体起源的内共生假说表明线粒体起源于特化的原核生物,线粒体DNA(mtDNA)和细菌基因组的相似性支持了这个概念【60】。mtDNA是否像细菌一样存在高水平的6mA,以及6mA在mtDNA中的功能引起了该领域的广泛关注。一些研究通过不同的方法检测mtDNA中的6mA水平显著高于核DNA。Koh等首次系统地研究了HEK293T细胞中的线粒体6mA。他们的质谱数据显示,与基因组DNA(<1 ppm)相比,mtDNA中的6mA水平明显更高(~18 ppm)【51】。在功能上,Koh等观察到在敲除6mA的去甲基化酶ALKBH1后,线粒体的氧化磷酸化减少。类似地,何川团队调查了多种细胞和组织中线粒体6mA的水平【45】。他们的质谱结果显示,在所有测试的样品中,6mA在mtDNA中明显富集,特别是在HepG2细胞中(在mtDNA中高达400ppm)。此外,他们在HepG2细胞中鉴定出了一种甲基转移酶(METTL4)。此外,何川团队还证明了6mA通过阻止TFAM与DNA结合来抑制线粒体基因转录。另外,何川团队发现缺氧可以进一步提高mtDNA中的6mA,表明线粒体6mA可能在调节缺氧应激反应中发挥重要作用。这些研究共同表明,与核DNA相比,6mA在mtDNA中的相对丰度高,而且mtDNA中的6mA在调节线粒体活性和应对像缺氧这样的环境压力方面起着关键作用。线粒体6mA和核内表观遗传调控之间是否存在联系是一个很有意思的问题。
6mA在DNA损伤修复中的作用
虽然6mA在DNA错配修复中的作用在原核生物中已得到证实,但6mA的这一作用在真核生物中是否保守仍不清楚。Zhang等人提出了一个的模型,即核苷酸切除修复(NER)位点的DNA 6mA可以防止8-氧代鸟嘌呤(8-oxoG)的错位,因为8-氧代鸟嘌呤错位到与未甲基化腺嘌呤相对的DNA中会导致T:A到G:C的转位突变【61】。因此,DNA 6mA可能作为一种保护性的DNA修饰,防止从T:A到G:C的转化。这个模型的一个问题是,核苷酸切除后的大多数单链腺嘌呤位点需要被甲基化,以有效防止8-氧代G的误入。考虑到复制错误率(约100,000分之一)和NER过程中单链DNA间隙的平均长度(约25-30 nt),6mA的丰度需要约62.5-75 ppm,以有效的防止8-oxoG的结合,这比以前报道的许多细胞系中6mA的基础水平(约0.1-7 ppm)至少高一个数量级。尽管如此,该模型在更特殊的条件下可能是成立的,如修复紫外线诱导的DNA损伤和双链断裂修复等。
在另一项研究中,汪海林团队报道 6mA在G1期积累,聚合酶λ(pol λ)可以直接将6mA整入mESCs的基因组中,表明6mA也可能在NHEJ途径中发挥作用【49】。然而,6mA在NHEJ通路中的确切作用仍然未知。需要进一步调查以更好地了解6mA在DNA损伤修复中的功能。
总的来说,尽管6mA已经被发现存在于多种真核生物当中,相比于经典的5mC, DNA 6mA在这些物种中的功能研究仍处于初始阶段。许多研究表明,与5mC相比,6mA在不同的环境压力下,包括缺氧、慢性应激,恐惧消退学习和线粒体应激受到的调节可能更为动态。这些发现表明,6mA可以作为一种表观遗传因子,对不同的环境压力作出快速反应,保护细胞免受更严重的后果。这些环境压力诱导6mA的上调的详细机制还需要进一步的研究。此外,研究者也应该针对这些特性来开发功能模型去探索6mA可能的甲基转移酶或阅读器。
结论和展望
近年来,DNA 6mA被发现存在于许多真核生物物种中。在功能上,6mA已被证实在调控基因表达、逆转录子抑制、应激反应、染色质组织、肿瘤发生和早期胚胎发育中发挥关键作用。关于6mA在哺乳动物基因组中的调控研究仍处于早期阶段。哺乳动物基因组中6mA的来源是通过DNA 甲基转移酶酶,如METTL4,还是来自于DNA聚合酶对甲基化碱基的错误整合,仍然需要更多的研究。6mA的去除是由ALKBH1、ALKBH4等去甲基酶介导的。虽然6mA已被证明可以阻止核小体和蛋白质(SATB1和TFAM)的DNA结合,但在哺乳动物中是否存在专门识别和结合6mA位点的 "阅读 "蛋白仍是一个开放的问题。因此,对哺乳动物中6mA的添加、去除和识别的机制途径进行更多的研究对于进一步了解其功能至关重要。特别是破译6mA在哺乳动物中的甲基转移酶仍然是最主要的挑战。
6mA研究的另一个主要挑战是难以在单碱基分辨率下准确检测6mA。我们相信,随着方法学的进步,6mA的检测方法很快会有突破性进展。这些新方法将极大地帮助研究人员更好地描述6mA在哺乳动物中的功能。
最后,科学家仍然需要寻找并建立确切的功能系统,才能加快6mA的研究步伐。
原文链接:
https://link.springer.com/content/pdf/10.1007/s13238-021-00866-3.pdf
休斯顿贝勒医学院吴涛实验室致力于研究衰老相关疾病,癌症发生,以及抗药性背后的表观遗传调控机理。欢迎有兴趣的人员加盟,可以直接联系,tao.wu@bcm.edu
简历投递(有意向者,请将个人简历等材料发至):
https://jinshuju.net/f/ZqXwZt
参考文献
[1] D. Schübeler, “Function and information content of DNA methylation,” Nature, vol. 517, no. 7534, pp. 321–326, Jan. 2015, doi: 10.1038/nature14192.
[2] Z. D. Smith and A. Meissner, “DNA methylation: roles in mammalian development,” Nat. Rev. Genet., vol. 14, no. 3, pp. 204–220, Mar. 2013, doi: 10.1038/nrg3354.
[3] C. Luo, P. Hajkova, and J. R. Ecker, “Dynamic DNA methylation: In the right place at the right time,” Science (80-. )., vol. 361, no. 6409, pp. 1336–1340, Sep. 2018, doi: 10.1126/science.aat6806.
[4] E. Li and Y. Zhang, “DNA methylation in mammals,” Cold Spring Harb. Perspect. Biol., vol. 6, no. 5, p. a019133, 2014, doi: 10.1101/cshperspect.a019133.
[5] Y. Fu et al., “N6-methyldeoxyadenosine marks active transcription start sites in Chlamydomonas,” Cell, vol. 161, no. 4, pp. 879–892, 2015, doi: 10.1016/j.cell.2015.04.010.
[6] G. Zhang et al., “N6-methyladenine DNA modification in Drosophila,” Cell, vol. 161, no. 4, pp. 893–906, 2015, doi: 10.1016/j.cell.2015.04.018.
[7] E. L. Greer et al., “DNA methylation on N6-adenine in C. elegans,” Cell, vol. 161, no. 4, pp. 868–878, 2015, doi: 10.1016/j.cell.2015.04.005.
[8] T. P. Wu et al., “DNA methylation on N6-adenine in mammalian embryonic stem cells,” Nature, vol. 532, no. 7599, pp. 329–333, Apr. 2016, doi: 10.1038/nature17640.
[9] D. B. Dunn and J. D. Smith, “Occurrence of a new base in the deoxyribonucleic acid of a strain of Bacterium coli,” Nature, vol. 175, no. 4451, pp. 336–337, 1955, doi: 10.1038/175336a0.
[10] D. B. Dunn and J. D. Smith, “The occurrence of 6-methylaminopurine in deoxyribonucleic acids,” Biochem. J., vol. 68, no. 4, pp. 627–636, Apr. 1958, doi: 10.1042/bj0680627.
[11] B. F. Vanyushin, A. N. Belozersky, N. A. Kokurina, and D. X. Kadirova, “5-methylcytosine and 6-methylaminopurine in bacterial DNA,” Nature, vol. 218, no. 5146, pp. 1066–1067, 1968, doi: 10.1038/2181066a0.
[12] H. W. Boyer, “DNA restriction and modification mechanisms in bacteria.,” Annu. Rev. Microbiol., vol. 25, no. 1, pp. 153–176, Oct. 1971, doi: 10.1146/annurev.mi.25.100171.001101.
[13] J. D. Smith, W. Arber, and U. Kühnlein, “Host specificity of DNA produced by Escherichia coli. XIV. the role of nucleotide methylation in in vivo B-specific modification,” J. Mol. Biol., vol. 63, no. 1, pp. 1–8, 1972, doi: 10.1016/0022-2836(72)90517-7.
[14] M. G. Marinus and N. R. Morris, “Isolation of deoxyribonucleic acid methylase mutants of Escherichia coli K-12,” J. Bacteriol., vol. 114, no. 3, pp. 1143–1150, Jun. 1973, doi: 10.1128/jb.114.3.1143-1150.1973.
[15] M. G. Marinus and N. R. Morris, “Biological function for 6-methyladenine residues in the DNA of Escherichia coli K12,” J. Mol. Biol., vol. 85, no. 2, pp. 309–322, 1974, doi: 10.1016/0022-2836(74)90366-0.
[16] M. Lu, J. L. Campbell, E. Boye, and N. Kleckner, “SeqA: A negative modulator of replication initiation in E. coli,” Cell, vol. 77, no. 3, pp. 413–426, May 1994, doi: 10.1016/0092-8674(94)90156-2.
[17] B. Kramer, W. Kramer, and H. J. Fritz, “Different base/base mismatches are corrected with different efficiencies by the methyl-directed DNA mismatch-repair system of E. coli,” Cell, vol. 38, no. 3, pp. 879–887, Oct. 1984, doi: 10.1016/0092-8674(84)90283-6.
[18] N. Sternberg, “Evidence that adenine methylation influences DNA-protein interactions in Escherichia coli,” J. Bacteriol., vol. 164, no. 1, pp. 490–493, Oct. 1985, doi: 10.1128/jb.164.1.490-493.1985.
[19] J. L. Robbins-Manke, Z. Z. Zdraveski, M. Marinus, and J. M. Essigmann, “Analysis of global gene expression and double-strand-break formation in DNA adenine methyltransferase- and mismatch repair-deficient Escherichia coli,” J. Bacteriol., vol. 187, no. 20, pp. 7027–7037, Oct. 2005, doi: 10.1128/JB.187.20.7027-7037.2005.
[20] M. A. Gorovsky, S. Hattman, and G. L. Pleger, “[6N]methyl adenine in the nuclear DNA of a eucaryote, Tetrahymena Pyriformis,” J. Cell Biol., vol. 56, no. 3, pp. 697–701, Mar. 1973, doi: 10.1083/jcb.56.3.697.
[21] S. Bromberg, K. Pratt, and S. Hattman, “Sequence specificity of DNA adenine methylase in the protozoan Tetrahymena thermophila,” J. Bacteriol., vol. 150, no. 2, pp. 993–996, May 1982, doi: 10.1128/jb.150.2.993-996.1982.
[22] J. Liu et al., “Abundant DNA 6mA methylation during early embryogenesis of zebrafish and pig,” Nat. Commun., vol. 7, p. 13052, 2016, doi: 10.1038/ncomms13052.
[23] C. Le Xiao et al., “N6 -Methyladenine DNA Modification in the Human Genome,” Mol. Cell, vol. 71, no. 2, pp. 306-318.e7, Jul. 2018, doi: 10.1016/j.molcel.2018.06.015.
[24] W. Huang, J. Xiong, Y. Yang, S. M. Liu, B. F. Yuan, and Y. Q. Feng, “Determination of DNA adenine methylation in genomes of mammals and plants by liquid chromatography/mass spectrometry,” RSC Adv., vol. 5, no. 79, pp. 64046–64054, Jul. 2015, doi: 10.1039/c5ra05307b.
[25] B. Liu, X. Liu, W. Lai, and H. Wang, “Metabolically Generated Stable Isotope-Labeled Deoxynucleoside Code for Tracing DNA N6-Methyladenine in Human Cells,” Anal. Chem., vol. 89, no. 11, pp. 6202–6209, Jun. 2017, doi: 10.1021/acs.analchem.7b01152.
[26] Z. K. O’Brown et al., “Sources of artifact in measurements of 6mA and 4mC abundance in eukaryotic genomic DNA,” BMC Genomics, vol. 20, no. 1, p. 445, Jun. 2019, doi: 10.1186/s12864-019-5754-6.
[27] Z. Li et al., “N6-methyladenine in DNA antagonizes SATB1 in early development,” Nature, vol. 583, no. 7817, pp. 625–630, Jul. 2020, doi: 10.1038/s41586-020-2500-9.
[28] K. Douvlataniotis, M. Bensberg, A. Lentini, B. Gylemo, and C. E. Nestor, “No evidence for DNA N6-methyladenine in mammals,” Sci. Adv., vol. 6, no. 12, p. eaay3335, 2020, doi: 10.1126/sciadv.aay3335.
[29] G. Z. Luo et al., “Characterization of eukaryotic DNA N6-methyladenine by a highly sensitive restriction enzyme-assisted sequencing,” Nat. Commun., vol. 7, p. 11301, 2016, doi: 10.1038/ncomms11301.
[30] G. Z. Luo et al., “N6-methyldeoxyadenosine directs nucleosome positioning in Tetrahymena DNA 06 Biological Sciences 0604 Genetics,” Genome Biol., vol. 19, no. 1, p. 200, 2018, doi: 10.1186/s13059-018-1573-3.
[31] Q. Xie et al., “N6-methyladenine DNA Modification in Glioblastoma,” Cell, vol. 175, no. 5, pp. 1228-1243.e20, 2018, doi: 10.1016/j.cell.2018.10.006.
[32] S. Zhu et al., “Mapping and characterizing N6-methyladenine in eukaryotic genomes using single-molecule real-time sequencing,” Genome Res., vol. 28, no. 7, pp. 1067–1078, 2018, doi: 10.1101/gr.231068.117.
[33] B. A. Flusberg et al., “Direct detection of DNA methylation during single-molecule, real-time sequencing,” Nat. Methods, vol. 7, no. 6, pp. 461–465, 2010, doi: 10.1038/nmeth.1459.
[34] Z. Liang et al., “DNA N6-Adenine Methylation in Arabidopsis thaliana,” Dev. Cell, vol. 45, no. 3, pp. 406-416.e3, 2018, doi: 10.1016/j.devcel.2018.03.012.
[35] C. Zhou et al., “Identification and analysis of adenine N6-methylation sites in the rice genome,” Nat. Plants, vol. 4, no. 8, pp. 554–563, 2018, doi: 10.1038/s41477-018-0214-x.
[36] A. C. Rand et al., “Mapping DNA methylation with high-throughput nanopore sequencing,” Nat. Methods, vol. 14, no. 4, pp. 411–413, 2017, doi: 10.1038/nmeth.4189.
[37] A. B. R. McIntyre et al., “Single-molecule sequencing detection of N6-methyladenine in microbial reference materials,” Nat. Commun., vol. 10, no. 1, p. 579, 2019, doi: 10.1038/s41467-019-08289-9.
[38] Q. Liu, L. Fang, G. Yu, D. Wang, C. Le Xiao, and K. Wang, “Detection of DNA base modifications by deep recurrent neural network on Oxford Nanopore sequencing data,” Nat. Commun., vol. 10, no. 1, p. 2449, 2019, doi: 10.1038/s41467-019-10168-2.
[39] P. Ni et al., “DeepSignal: Detecting DNA methylation state from Nanopore sequencing reads using deep-learning,” Bioinformatics, vol. 35, no. 22, pp. 4586–4595, 2019, doi: 10.1093/bioinformatics/btz276.
[40] T. Hong et al., “Selective detection of N6-methyladenine in DNA via metal ion-mediated replication and rolling circle amplification,” Chem. Sci., vol. 8, no. 1, pp. 200–205, 2016, doi: 10.1039/C6SC02271E.
[41] M. Nappi, A. Hofer, S. Balasubramanian, and M. J. Gaunt, “Selective Chemical Functionalization at N6-Methyladenosine Residues in DNA Enabled by Visible-Light-Mediated Photoredox Catalysis,” J. Am. Chem. Soc., vol. 142, no. 51, pp. 21484–21492, Dec. 2020, doi: 10.1021/jacs.0c10616.
[42] Y. Mahdavi-Amiri, K. Chung Kim Chung, and R. Hili, “Single-nucleotide resolution of: N6-adenine methylation sites in DNA and RNA by nitrite sequencing,” Chem. Sci., vol. 12, no. 2, pp. 606–612, 2021, doi: 10.1039/d0sc03509b.
[43] S. M. Kweon, Y. Chen, E. Moon, K. Kvederaviciutė, S. Klimasauskas, and D. E. Feldman, “An Adversarial DNA N6-Methyladenine-Sensor Network Preserves Polycomb Silencing,” Mol. Cell, vol. 74, no. 6, pp. 1138-1147.e6, 2019, doi: 10.1016/j.molcel.2019.03.018.
[44] W. Li, Y. Shi, T. Zhang, J. Ye, and J. Ding, “Structural insight into human N6amt1–Trm112 complex functioning as a protein methyltransferase,” Cell Discov., vol. 5, no. 1, pp. 1–13, 2019, doi: 10.1038/s41421-019-0121-y.
[45] Z. Hao et al., “N6-Deoxyadenosine Methylation in Mammalian Mitochondrial DNA,” Mol. Cell, vol. 78, no. 3, pp. 382-395.e8, 2020, doi: 10.1016/j.molcel.2020.02.018.
[46] C. B. Woodcock et al., “Human MettL3–MettL14 complex is a sequence-specific DNA adenine methyltransferase active on single-strand and unpaired DNA in vitro,” Cell Discov., vol. 5, no. 1, p. 63, 2019, doi: 10.1038/s41421-019-0136-4.
[47] M. P. Charles et al., “N6-Methyldeoxyadenosine, a nucleoside commonly found in prokaryotes, induces C2C12 myogenic differentiation,” Biochem. Biophys. Res. Commun., vol. 314, no. 2, pp. 476–482, 2004, doi: 10.1016/j.bbrc.2003.12.132.
[48] M. U. Musheev, A. Baumgärtner, L. Krebs, and C. Niehrs, “The origin of genomic N 6-methyl-deoxyadenosine in mammalian cells,” Nat. Chem. Biol., vol. 16, no. 6, pp. 630–634, 2020, doi: 10.1038/s41589-020-0504-2.
[49] X. Liu et al., “N6-methyladenine is incorporated into mammalian genome by DNA polymerase,” Cell Res., vol. 31, no. 1, pp. 94–97, 2021, doi: 10.1038/s41422-020-0317-6.
[50] S. He et al., “6mA-DNA-binding factor Jumu controls maternal-to-zygotic transition upstream of Zelda,” Nat. Commun., vol. 10, no. 1, pp. 1–14, 2019, doi: 10.1038/s41467-019-10202-3.
[51] C. W. Q. Koh et al., “Single-nucleotide-resolution sequencing of human N6-methyldeoxyadenosine reveals strand-asymmetric clusters associated with SSBP1 on the mitochondrial genome,” Nucleic Acids Res., vol. 46, no. 22, pp. 11659–11670, 2018, doi: 10.1093/nar/gky1104.
[52] M. Zhang et al., “Mammalian ALKBH1 serves as an N6-mA demethylase of unpairing DNA,” Cell Res., vol. 30, no. 3, pp. 197–210, 2020, doi: 10.1038/s41422-019-0237-5.
[53] L.-F. Tian et al., “Structural basis of nucleic acid recognition and 6mA demethylation by human ALKBH1.,” Cell Res., vol. 30, no. 3, pp. 272–275, Mar. 2020, doi: 10.1038/s41422-019-0233-9.
[54] H. Li et al., “ALKBH1 deficiency leads to loss of homeostasis in human diploid somatic cells,” Protein Cell, vol. 11, no. 9, pp. 688–695, Sep. 2020, doi: 10.1007/s13238-020-00744-4.
[55] B. Yao et al., “DNA N6-methyladenine is dynamically regulated in the mouse brain following environmental stress,” Nat. Commun., vol. 8, no. 1, p. 1122, 2017, doi: 10.1038/s41467-017-01195-y.
[56] X. Li et al., “The DNA modification N6-methyl-2’-deoxyadenosine (m6dA) drives activity-induced gene expression and is required for fear extinction,” Nat. Neurosci., vol. 22, no. 4, pp. 534–544, Apr. 2019, doi: 10.1038/s41593-019-0339-x.
[57] M. Kulis and M. Esteller, “DNA Methylation and Cancer,” Adv. Genet., vol. 70, no. C, pp. 27–56, 2010, doi: 10.1016/B978-0-12-380866-0.60002-2.
[58] L. Chen, M. Zhang, and M. Guo, “DNA N6-methyladenine Epigenetic Modification Elevated in Human Esophageal Squamous Cell Carcinoma: a Potential Prognostic Marker,” Discov. Med., vol. 157, no. 29, pp. 85–90, 2020.
[59] D. Liang et al., “The decreased N6-methyladenine DNA modification in cancer cells,” Biochem. Biophys. Res. Commun., vol. 480, no. 1, pp. 120–125, 2016, doi: 10.1016/j.bbrc.2016.09.136.
[60] M. W. Gray, “Mitochondrial evolution,” Cold Spring Harb. Perspect. Biol., vol. 4, no. 9, p. a011403, Sep. 2012, doi: 10.1101/cshperspect.a011403.
[61] X. Zhang, R. M. Blumenthal, and X. Cheng, “A Role for N6-Methyladenine in DNA Damage Repair,” Trends Biochem. Sci., vol. 46, no. 3, pp. 175–183, 2021, doi: 10.1016/j.tibs.2020.09.007.
(可上下滑动阅览)
来源:BioArt
网站:www.cscb.org.cn
微信:CSCB-WeChat
原标题:《【重磅综述】 “路漫漫其修远兮,吾将上下而求索”,DNA 6mA修饰研究全纪录》
本文为澎湃号作者或机构在澎湃新闻上传并发布,仅代表该作者或机构观点,不代表澎湃新闻的观点或立场,澎湃新闻仅提供信息发布平台。申请澎湃号请用电脑访问http://renzheng.thepaper.cn。





- 报料热线: 021-962866
- 报料邮箱: news@thepaper.cn
互联网新闻信息服务许可证:31120170006
增值电信业务经营许可证:沪B2-2017116
© 2014-2024 上海东方报业有限公司




